はじめに
『ベイズ推論による機械学習入門』の学習時のノートです。基本的な内容は「数式の行間を読んでみた」とそれを「RとPythonで組んでみた」になります。「数式」と「プログラム」から理解するのが目標です。
この記事は、4.3.2項の内容です。「観測モデルをポアソン混合分布」、「事前分布をガンマ分布とディリクレ分布」とするポアソン混合モデルに対するギブスサンプリングをPythonで実装します。
省略してある内容等ありますので、本とあわせて読んでください。初学者な自分が理解できるレベルまで落として書き下していますので、分かる人にはかなりくどくなっています。同じような立場の人のお役に立てれば幸いです。
【数式読解編】
【他の節の内容】
【この節の内容】
・Pythonでやってみる
人工的に生成したデータを用いて、ベイズ推論を行ってみましょう。アルゴリズム4.2を参考に実装します。
利用するライブラリを読み込みます。
# 4.3.2項で利用するライブラリ import numpy as np from scipy.stats import poisson, gamma # ポアソン分布, ガンマ分布 import matplotlib.pyplot as plt
・モデルの設定
まずは、観測モデルを設定します。この例では、$K$個の観測モデルをポアソン分布$\mathrm{Poi}(x_n | \lambda_k)$とします。また、各データに対する$K$個の観測モデルの割り当てをカテゴリ分布$\mathrm{Cat}(\mathbf{s}_n | \boldsymbol{\pi})$によって行います。
観測モデルのパラメータと混合比率を設定します。
# K個の真のパラメータを指定 lambda_truth_k = np.array([10.0, 25.0, 40.0]) # 真の混合比率を指定 pi_truth_k = np.array([0.35, 0.25, 0.4]) # クラスタ数を取得 K = len(lambda_truth_k)
観測モデルの$K$個のパラメータ$\boldsymbol{\lambda} = \{\lambda_1, \lambda_2, \cdots, \lambda_K\}$をlambda_truth_kとして、$\lambda_k \geq 0$の値を指定します。
混合比率パラメータ$\boldsymbol{\pi} = (\pi_1, \pi_2, \cdots, \pi_K)$をpi_truth_kとして、$\sum_{k=1}^K \pi_k = 1$、$\pi_k \geq 0$の値を指定します。
この2つのパラメータの値を求めるのがここでの目的です。
設定したモデルをグラフで確認しましょう。グラフ用の点を作成します。
# 作図用のxの点を作成 x_line = np.arange(0, 2 * np.max(lambda_truth_k)) # 確認 print(x_line[:10])
[0. 1. 2. 3. 4. 5. 6. 7. 8. 9.]
作図用に、ポアソン分布に従う変数$x_n$がとり得る非負の整数をnp.arange()で作成してx_lineとします。この例では、0から$\boldsymbol{\lambda}$の最大値の2倍を範囲とします。
ポアソン混合分布の確率を計算します。
# 観測モデルを計算 model_prob = 0.0 for k in range(K): # クラスタkの分布の確率を計算 tmp_prob = poisson.pmf(k=x_line, mu=lambda_truth_k[k]) # K個の分布の加重平均を計算 model_prob += pi_truth_k[k] * tmp_prob
$K$個のパラメータlambda_truth_kを使って、x_lineの値ごとに次の式で確率を計算します。
$K$個のポアソン分布に対して、$\boldsymbol{\pi}$を使って加重平均をとります。
ポアソン分布の確率は、SciPyライブラリのstatsモジュールのpoisson.pmf()で計算できます。データの引数kにx_line、パラメータの引数muにlambda_truth_k[k]を指定します。
この計算をfor文でK回繰り返し行います。各パラメータによって求めた$K$回分の確率をpi_truth_k[k]で割り引いてmodel_probに加算していきます。
計算結果は次のようになります。
# 確認 print(np.round(model_prob[:10], 3))
[0. 0. 0.001 0.003 0.007 0.013 0.022 0.032 0.039 0.044]
観測モデルを作図します。
# 観測モデルを作図 plt.figure(figsize=(12, 9)) plt.bar(x=x_line, height=model_prob) # 真の分布 plt.xlabel('x') plt.ylabel('prob') plt.suptitle('Poisson Mixture Model', size = 20) plt.title('$\lambda=[' + ', '.join([str(lmd) for lmd in lambda_truth_k]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in pi_truth_k])+ ']$', loc='left') plt.show()
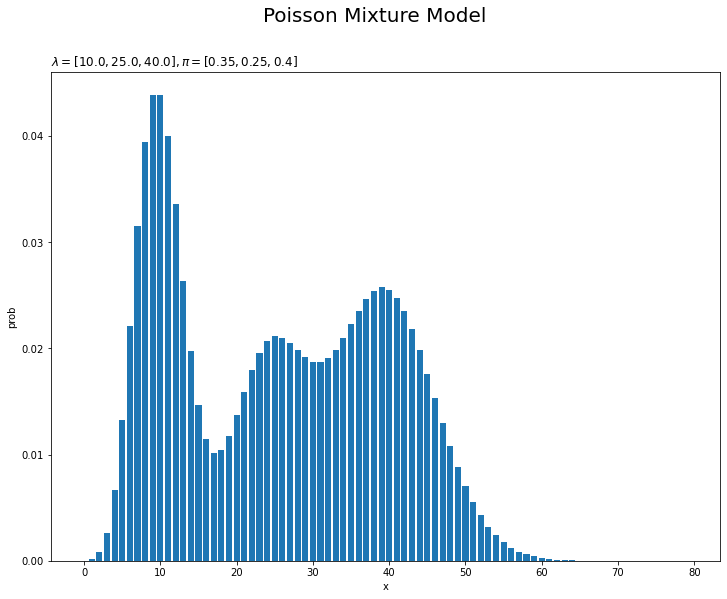
$K$個の分布を重ねたような分布になります。ただし、総和が1となるようにしているため、$K$個の分布そのものよりも1つ1つの山が小さく(確率値が小さく)なっています。
・データの生成
続いて、構築したモデルに従ってデータを生成します。先に、潜在変数$\mathbf{S} = \{\mathbf{s}_1, \mathbf{s}_2, \cdots, \mathbf{s}_N\}$、$\mathbf{s}_n = (s_{n,1}, s_{n,2}, \cdots, s_{n,K})$を生成し、各データに割り当てられたクラスタに従い観測データ$\mathbf{X} = \{x_1, x_2, \cdots, x_N\}$を生成します。
$N$個の潜在変数$\mathbf{S}$を生成します。
# (観測)データ数を指定 N = 250 # 真のクラスタを生成 s_truth_nk = np.random.multinomial(n=1, pvals=pi_truth_k, size=N)
生成するデータ数$N$をNとして値を指定します。
カテゴリ分布に従う乱数は、多項分布に従う乱数生成関数np.random.multinomial()のn引数を1にすることで生成できます。また、確率(パラメータ)の引数pvalsにpi_truth_k、試行回数の引数sizeにNを指定します。生成した$N$個の$K$次元ベクトルをs_truth_nkとします。
結果は次のようになります。
# 確認 print(s_truth_nk[:5])
[[0 1 0]
[1 0 0]
[0 1 0]
[0 1 0]
[1 0 0]]
これが本来は観測できない潜在変数であり、真のクラスタです。各行がデータを表し、値が1の列番号が割り当てられたクラスタ番号を表します。
s_truth_nkから各データのクラスタ番号を抽出します。
# 真のクラスタ番号を抽出 _, s_truth_n = np.where(s_truth_nk == 1) # 確認 print(s_truth_n[:5])
[1 0 1 1 0]
np.where()を使って行(データ)ごとに要素が1の列番号を抽出します。
各データのクラスタに従い$N$個のデータを生成します。
# (観測)データを生成
x_n = np.random.poisson(lam=lambda_truth_k[s_truth_n], size=N)
ポアソン分布に従う乱数は、np.random.poisson()で生成できます。パラメータの引数lamにlambda_truth_k[s_truth_n]、試行回数の引数sizeにNを指定します。各データのクラスタ番号s_truth_nを添字に使うことで、各データのクラスタに対応したパラメータ$\lambda_k$をlambda_truth_kから取り出すことができます。生成した$N$個のデータをx_nとします。式(4.28)を再現して、lamにapply(lambda_truth_k^t(s_truth_nk), 2, prod)を渡しても生成できます。
観測したデータを確認します。
# 確認 print(x_n[:10])
[32 11 27 22 11 22 21 13 13 46]
観測データが非負の整数になっているのを確認できます。
観測データをヒストグラムで確認しましょう。
# 観測データのヒストグラムを作成 plt.figure(figsize=(12, 9)) plt.bar(x=x_line, height=model_prob, label='true model', color='white', alpha=1, edgecolor='red', linestyle='--') # 真の分布 plt.bar(x=x_line, height=[np.sum(x_n == x) / len(x_n) for x in x_line], label='observation data') # 観測データ plt.xlabel('x') plt.ylabel('dens') plt.suptitle('Poisson Mixture Model', fontsize=20) plt.title('$N=' + str(N) + ', \lambda=[' + ', '.join([str(lmd) for lmd in lambda_truth_k]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in pi_truth_k]) + ']$', loc='left') plt.legend() plt.show()
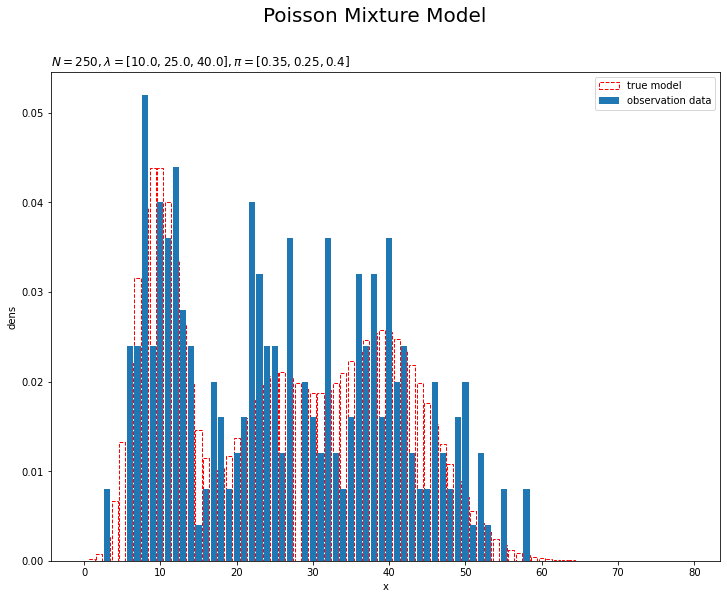
真の分布を重ねて描画しています。
また、クラスタもヒストグラムで確認しましょう。
# 真のクラスタのヒストグラムを作成 plt.figure(figsize=(12, 9)) for k in range(K): plt.bar(x=x_line, height=[np.sum(x_n[s_truth_n == k] == x) for x in x_line], alpha=0.5, label='cluster:' + str(k + 1)) # 真のクラスタ plt.xlabel('x') plt.ylabel('count') plt.suptitle('Poisson Mixture Model', fontsize=20) plt.title('$N=' + str(N) + ', \lambda=[' + ', '.join([str(lmd) for lmd in lambda_truth_k]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in pi_truth_k]) + ']$', loc='left') plt.legend() plt.show()

どちらの分布もデータが十分に大きいと真の分布に近づきます。
・事前分布の設定
次に、観測モデル(観測データの分布)$p(\mathbf{X} | \boldsymbol{\lambda})$と潜在変数の分布$p(\mathbf{S} | \boldsymbol{\pi})$に対する共役事前分布を設定します。ポアソン分布のパラメータ$\boldsymbol{\lambda}$に対する事前分布$p(\boldsymbol{\lambda})$としてガンマ分布$\mathrm{Gam}(\boldsymbol{\lambda} | a, b)$、カテゴリ分布のパラメータ$\boldsymbol{\pi}$に対する事前分布$p(\boldsymbol{\pi})$としてディリクレ分布$p(\boldsymbol{\pi} | \boldsymbol{\alpha})$を設定します。
$\boldsymbol{\lambda}$の事前分布と$\boldsymbol{\pi}$の事前分布のパラメータ(超パラメータ)を設定します。
# lambdaの事前分布のパラメータを指定 a = 1.0 b = 1.0 # piの事前分布のパラメータを指定 alpha_k = np.repeat(2.0, K)
ガンマ分布のパラメータ$a,\ b$をそれぞれa, bとして、$a > 0,\ b > 0$の値を指定します。
ディリクレ分布のパラメータ$\boldsymbol{\alpha} = (\alpha_1, \alpha_2, \cdots, \alpha_K)$をalpha_kとして、$\alpha_k > 0$の値を指定します。
超パラメータを設定できたので、$\boldsymbol{\lambda}$の事前分布を確認しましょう。グラフ用の点を作成して、確率密度を計算します。
# 作図用のlambdaの点を作成 lambda_line = np.linspace(0.0, 2.0 * np.max(lambda_truth_k), num=1000) # lambdaの事前分布を計算 prior_lambda = gamma.pdf(x=lambda_line, a=a, scale=1 / b)
ガンマ分布に従う変数$\lambda_k$がとり得る0以上の値をnp.linspace()で作成してlambda_lineとします。この例では、0から$\boldsymbol{\lambda}$の最大値の2倍を範囲とします。np.linspace()を使うと指定した要素数で等間隔に切り分けます。np.arange()を使うと切り分ける間隔を指定できます。処理が重い場合は、この値を調整してください。
作成したlambda_lineの値ごとに確率密度を計算します。ガンマ分布の確率密度は、SciPyライブラリのstatsモジュールのgamma.pdf()で計算できます。データの引数xにlambda_line、a引数にa、scale引数にbを指定します。
計算結果は次のようになります。
# 確認 print(np.round(lambda_line[:10], 2)) print(np.round(prior_lambda[:10], 2))
[0. 0.08 0.16 0.24 0.32 0.4 0.48 0.56 0.64 0.72]
[1. 0.92 0.85 0.79 0.73 0.67 0.62 0.57 0.53 0.49]
$\boldsymbol{\lambda}$の事前分布を作図します。
# lambdaの事前分布を作図 plt.figure(figsize=(12, 9)) plt.plot(lambda_line, prior_lambda, label='prior', color='purple') # 事前分布 plt.vlines(x=lambda_truth_k, ymin=0.0, ymax=np.max(prior_lambda), label='true val', color='red', linestyle='--') # 真の値 plt.xlabel('$\lambda$') plt.ylabel('density') plt.suptitle('Gamma Distribution', fontsize=20) plt.title('a=' + str(a) + ', b=' + str(b), loc='left') plt.legend() plt.show()

真の値を垂直線plt.vlines()で描画しています。
$\pi$の事前分布(と事後分布)については省略します。詳しくは、3.2.2項「カテゴリ分布の学習と予測」をご参照ください。
・初期値の設定
設定した事前分布に従いパラメータ$\boldsymbol{\lambda},\ \boldsymbol{\pi}$を生成して初期値とします。
それぞれ事前分布に従い$\boldsymbol{\lambda}$と$\boldsymbol{\pi}$をサンプルします。
# lambdaを生成 lambda_k = np.random.gamma(shape=a, scale=1 / b, size=K) # piを生成 pi_k = np.random.dirichlet(alpha=alpha_k, size=1).reshape(K)
ガンマ分布に従う乱数はnp.random.gamma()で生成できます。データ数の引数sizeにKを指定します。他の引数については、gamma.pdf()と同じです。生成した$K$個のパラメータをlambda_kとします。
ディリクレ分布に従う乱数は、np.random.dirichlet()で生成できます。2次元配列で出力されるため、reshape(K)で1次元配列に変換しておきます。
生成したパラメータを確認します。
# 確認 print(lambda_k) print(pi_k)
[0.35550085 0.17254374 2.04555575]
[0.27973663 0.52242674 0.19783663]
パラメータの初期値を使った分布を確認します。
# 初期値による混合分布を計算 init_prob = 0.0 for k in range(K): # クラスタkの分布の確率を計算 tmp_prob = poisson.pmf(k=x_line, mu=lambda_k[k]) # K個の分布の加重平均を計算 init_prob += tmp_prob * pi_k[k] # 初期値による分布を作図 plt.figure(figsize=(12, 9)) plt.bar(x=x_line, height=model_prob, label='true model', color='white', alpha=1, edgecolor='red', linestyle='--') # 真の分布 plt.bar(x_line, init_prob, color='purple') # 初期値による分布 plt.xlabel('x') plt.ylabel('prob') plt.suptitle('Poisson Mixture Model', size = 20) plt.title('$iter:' + str(0) + ', \lambda=[' + ', '.join([str(lmd) for lmd in np.round(lambda_k, 2)]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in np.round(pi_k, 2)]) + ']$', loc='left') plt.legend() plt.show()
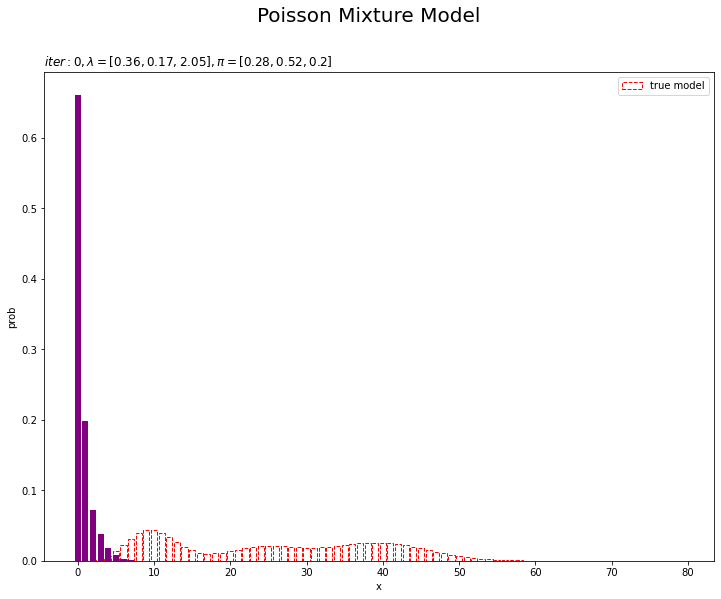
観測モデルのときと同様に作図します。
・ギブスサンプリング
ギブスサンプリングによりパラメータと潜在変数を推定します。
・コード全体
eta_nkとs_nkについては添字を使って代入するため、予め変数を用意しておく必要があります。
また、後で各パラメータの更新値や分布の推移を確認するために、それぞれ試行ごとにtrace_***に値を保存していきます。
# 試行回数を指定 MaxIter = 100 # 受け皿を作成 eta_nk = np.empty((N, K)) s_nk = np.empty((N, K)) # 推移の確認用の受け皿を作成 trace_s_in = [[np.nan] * N] trace_a_ik = [[a] * K] trace_b_ik = [[b] * K] trace_alpha_ik = [list(alpha_k)] trace_lambda_ik = [list(lambda_k)] trace_pi_ik = [list(pi_k)] # ギブスサンプリング for i in range(MaxIter): for n in range(N): # 潜在変数の事後分布のパラメータを計算:式(4.38) tmp_eta_k = np.exp(x_n[n] * np.log(lambda_k) - lambda_k + np.log(pi_k)) eta_nk[n] = tmp_eta_k / np.sum(tmp_eta_k) # 正規化 # クラスタをサンプル:式(4.37) s_nk[n] = np.random.multinomial(n=1, pvals=eta_nk[n]) # lambdaの事後分布のパラメータを計算:式(4.42) a_hat_k = np.sum(s_nk.T * x_n, axis=1) + a b_hat_k = np.sum(s_nk, axis=0) + b # lambdaをサンプル:式(4.41) lambda_k = np.random.gamma(shape=a_hat_k, scale=1 / b_hat_k, size=K) # piの事後分布のパラメータを計算:式(4.45) alpha_hat_k = np.sum(s_nk, axis=0) + alpha_k # piをサンプル:式(4.44) pi_k = np.random.dirichlet(alpha=alpha_hat_k, size=1).reshape(K) # 値を記録 _, s_n = np.where(s_nk == 1) # クラスタ番号を抽出 trace_s_in.append(list(s_n)) trace_a_ik.append(list(a_hat_k)) trace_b_ik.append(list(b_hat_k)) trace_alpha_ik.append(list(alpha_hat_k)) trace_lambda_ik.append(list(lambda_k)) trace_pi_ik.append(list(pi_k)) # 動作確認 #print(str(i+1) + ' (' + str(np.round((i + 1) / MaxIter * 100, 1)) + '%)')
・処理の確認
続いて、ギブスサンプリングで行う処理を確認していきます。
・潜在変数$\mathbf{S}$のサンプリング
$\mathbf{s}_n$の条件付き分布(事後分布)$p(\mathbf{s}_n | \mathbf{X}, \boldsymbol{\lambda}, \boldsymbol{\pi})$のパラメータを計算して、新たな$\mathbf{s}_n$を生成します。
for n in range(N): # 潜在変数の事後分布のパラメータを計算:式(4.38) tmp_eta_k = np.exp(x_n[n] * np.log(lambda_k) - lambda_k + np.log(pi_k)) eta_nk[n] = tmp_eta_k / np.sum(tmp_eta_k) # 正規化 # クラスタをサンプル:式(4.37) s_nk[n] = np.random.multinomial(n=1, pvals=eta_nk[n])
$n$番目のデータの潜在変数$\mathbf{s}_n$の事後分布(カテゴリ分布)のパラメータ$\boldsymbol{\eta}_n = (\eta_{n,1}, \eta_{n,2}, \cdots, \eta_{n,K})$の各項を次の式で計算します。
右辺の計算結果をtmp_eta_kとします。さらに、総和が1になるように
で正規化して、結果をeta_nkとします。
ちなみに、式(4.38)の$\exp(\cdot)$の括弧の中を$\tilde{\eta}_{n,k}$とすると、正規化の式は$\frac{\exp (\tilde{\eta}_{n,k})}{\sum_{k'=1}^K \exp (\tilde{\eta}_{n,k'})}$となります。これはソフトマックス関数の計算になります。オーバーフローが起きる場合は、「ソフトマックス関数のオーバーフロー対策【ゼロつく1のノート(数学)】 - からっぽのしょこ」を参考にしてください。
更新したパラメータeta_nk[n, ]を持つカテゴリ分布に従い$\boldsymbol{s}_n$をサンプルします。
結果は次のようになります。
# 確認 print(np.round(eta_nk[n], 5)) print(s_nk[n])
[0.00288 0.99712 0. ]
[0. 1. 0.]
この処理をfor文で$N$回繰り返すことで、全ての潜在変数のサンプルが得られます。
更新した$\mathbf{S}$を用いて、パラメータ$\boldsymbol{\lambda},\ \boldsymbol{\pi}$を更新します。
・観測モデルのパラメータ$\boldsymbol{\lambda}$のサンプリング
$\boldsymbol{\lambda}$の条件付き分布(事後分布)$p(\boldsymbol{\lambda} | \mathbf{X}, \mathbf{S})$のパラメータを計算して、新たな$\boldsymbol{\lambda}$を生成します。
# lambdaの事後分布のパラメータを計算:式(4.42) a_hat_k = np.sum(s_nk.T * x_n, axis=1) + a b_hat_k = np.sum(s_nk, axis=0) + b # lambdaをサンプル:式(4.41) lambda_k = np.random.gamma(shape=a_hat_k, scale=1 / b_hat_k, size=K)
$\boldsymbol{\lambda}$の条件付き分布($K$個のガンマ分布)のパラメータ$\hat{\mathbf{a}} = \{\hat{a}_1, \hat{a}_2, \cdots, \hat{a}_K\}$、$\hat{\mathbf{b}} = \{\hat{b}_1, \hat{b}_2, \cdots, \hat{b}_K\}$の各項を
で計算して、結果をa_hat_k, b_hat_kとします。
a_hat_k, b_hat_kを用いて、新たな$\boldsymbol{\lambda}$をサンプルします。
結果は次のようになります。
# 確認 print(a_hat_k) print(b_hat_k) print(lambda_k)
[1827. 819. 3956.]
[75. 81. 97.]
[23.65455328 9.78443817 40.43214946]
ちなみに、$\hat{a}_k$の計算式に関して、$s_{n,k} x_n$の計算は
print(x_n[:5]) print((s_nk[:5].T * x_n[:5]).T)
[32 11 27 22 11]
[[ 0. 0. 32.]
[ 0. 11. 0.]
[27. 0. 0.]
[22. 0. 0.]
[ 0. 11. 0.]]
各観測データをクラスタごとに仕分ける操作であり、$\sum_{n=1}^N s_{n,k} x_n$は
print(np.sum(s_nk[:5].T * x_n[:5], axis=1))
[49. 22. 32.]
割り当てられたクラスタごとに観測データの値の和をとる操作と言えます。また、$\sum_{n=1}^N s_{n,k}$の計算は
print(s_nk[:5]) print(np.sum(s_nk[:5], axis=0))
[[0. 0. 1.]
[0. 1. 0.]
[1. 0. 0.]
[1. 0. 0.]
[0. 1. 0.]]
[2. 2. 1.]
各クラスタを割り当てられたデータ数です。
・混合比率パラメータ$\boldsymbol{\pi}$のサンプリング
$\boldsymbol{\pi}$の条件付き分布(事後分布)$p(\boldsymbol{\pi} | \mathbf{X}, \mathbf{S})$のパラメータを計算して、新たな$\boldsymbol{\pi}$を生成します。
# piの事後分布のパラメータを計算:式(4.45) alpha_hat_k = np.sum(s_nk, axis=0) + alpha_k # piをサンプル:式(4.44) pi_k = np.random.dirichlet(alpha=alpha_hat_k, size=1).reshape(K)
$\boldsymbol{\pi}$の条件付き分布(ディリクレ分布)のパラメータ$\hat{\boldsymbol{\alpha}} = (\hat{\alpha}_1, \hat{\alpha}_2, \cdots, \hat{\alpha}_K)$の各項を
で計算して、結果をalpha_hat_kとします。
alpha_hat_kを用いて、新たな$\boldsymbol{\pi}$をサンプルします。
$\boldsymbol{\lambda},\ \boldsymbol{\pi}$が得られました。今度は、更新した$\boldsymbol{\lambda},\ \boldsymbol{\pi}$を用いて、潜在変数$\mathbf{S}$を更新します。この処理を指定した回数繰り返し行います。
・推論結果の確認
・最後のパラメータの確認
超パラメータa_hat_kとb_hat_kの最後の更新値を用いて、$\boldsymbol{\lambda}$の事後分布を計算します。
# lambdaの事後分布を計算 posterior_lambda_k = np.empty((K, len(lambda_line))) for k in range(K): posterior_lambda_k[k] = gamma.pdf(x=lambda_line, a=a_hat_k[k], scale=1 / b_hat_k[k])
$K$個のガンマ分布を計算して、データフレームに格納していきます。
作成したデータフレームは次のようになります。
# 確認 print(posterior_lambda_k)
[[0. 0. 0. ... 0. 0. 0.]
[0. 0. 0. ... 0. 0. 0.]
[0. 0. 0. ... 0. 0. 0.]]
$\boldsymbol{\lambda}$の事後分布を作図します。
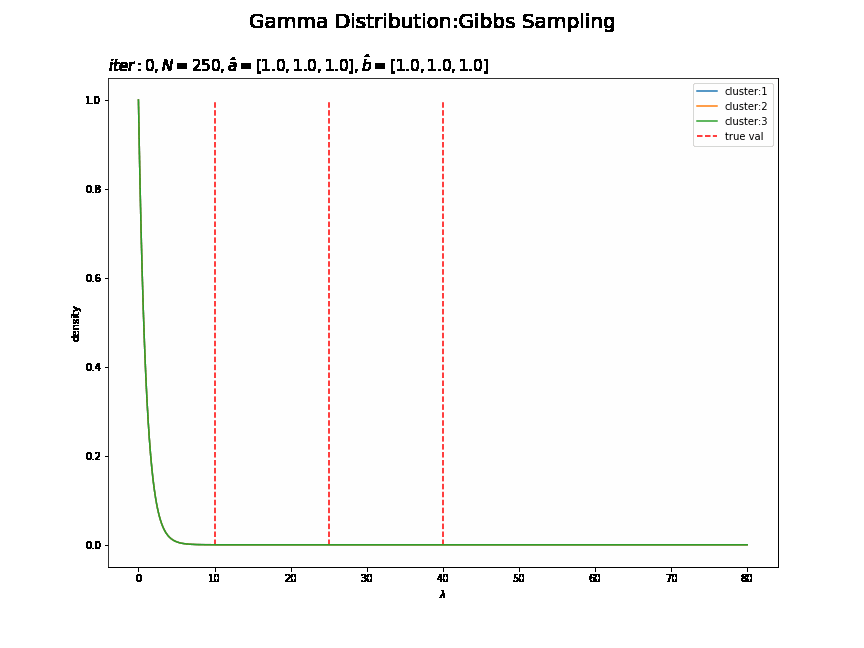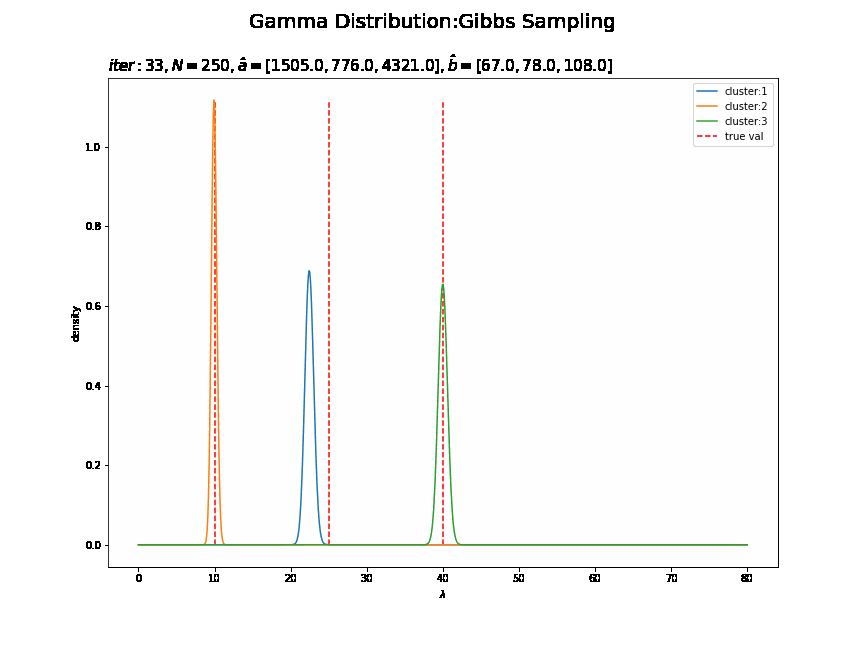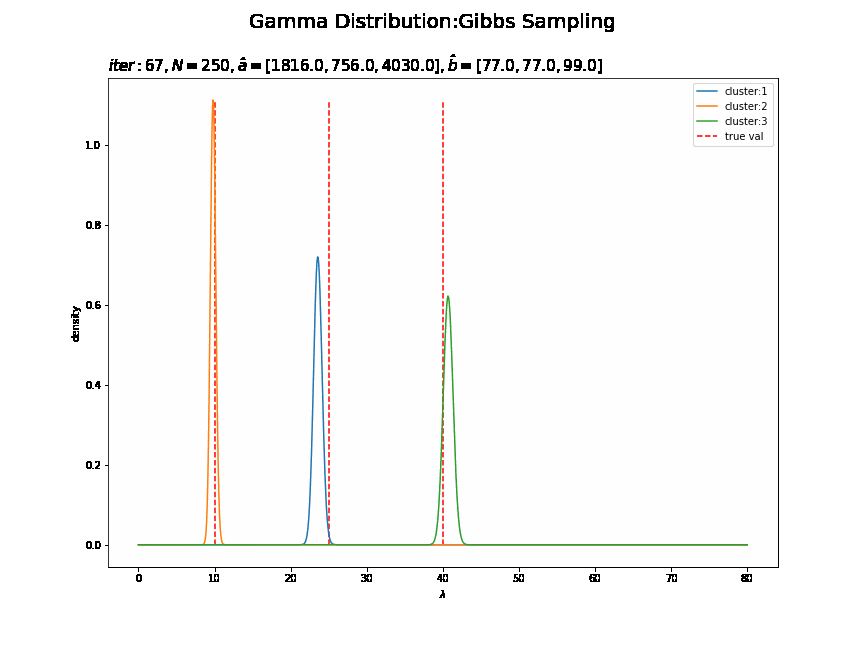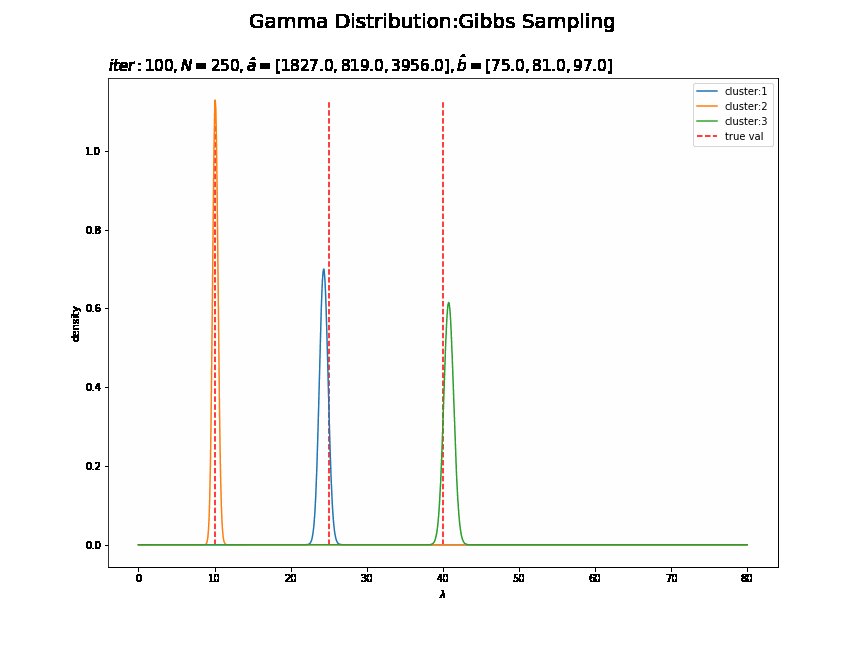
# lambdaの事後分布を作図 plt.figure(figsize=(12, 9)) for k in range(K): plt.plot(lambda_line, posterior_lambda_k[k], label='cluster:' + str(k + 1)) # 事後分布 plt.vlines(x=lambda_truth_k, ymin=0.0, ymax=np.max(posterior_lambda_k), label='true val', color='red', linestyle='--') # 真の値 plt.xlabel('$\lambda$') plt.ylabel('density') plt.suptitle('Gamma Distribution', fontsize=20) plt.title('$iter:' + str(MaxIter) + ', N=' + str(N) + ', \hat{a}=[' + ', '.join([str(a) for a in a_hat_k]) + ']' + ', \hat{b}=[' + ', '.join([str(b) for b in b_hat_k]) + ']$', loc='left') plt.legend() plt.show()
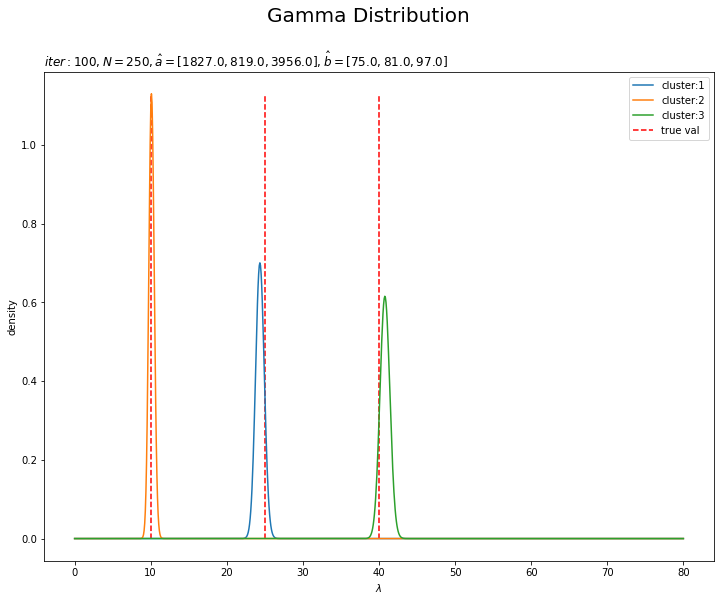
事前分布のときと同様に作図できます。
3つのガンマ分布がそれぞれ3つの真の値付近をピークとする分布を推定できています。
パラメータlambda_kとpi_kの最後のサンプルを用いて、混合分布を計算します。
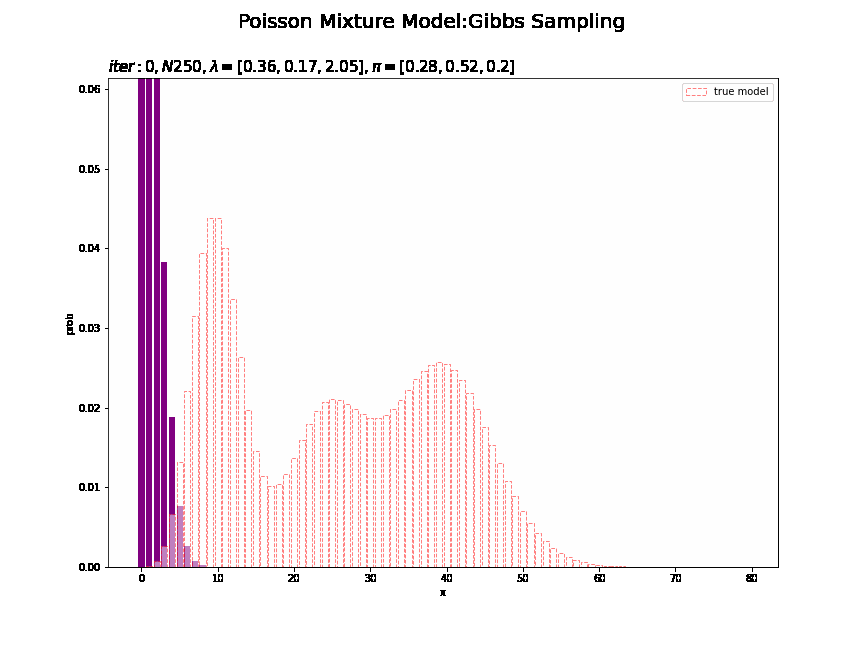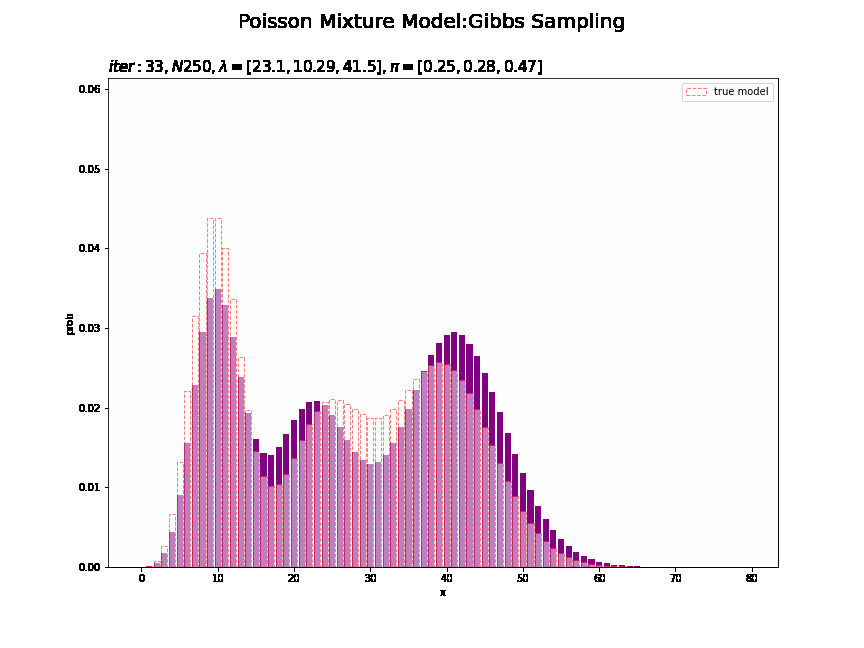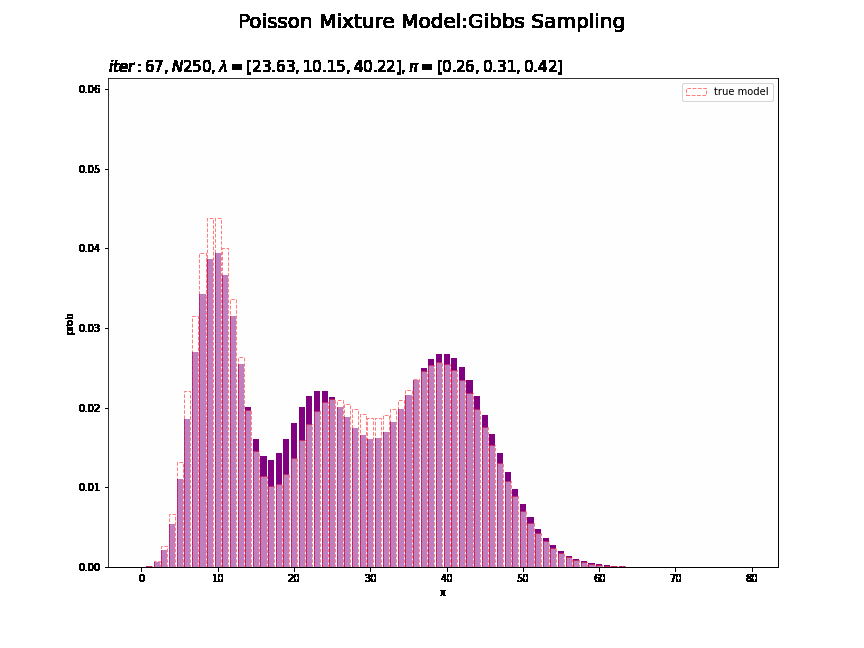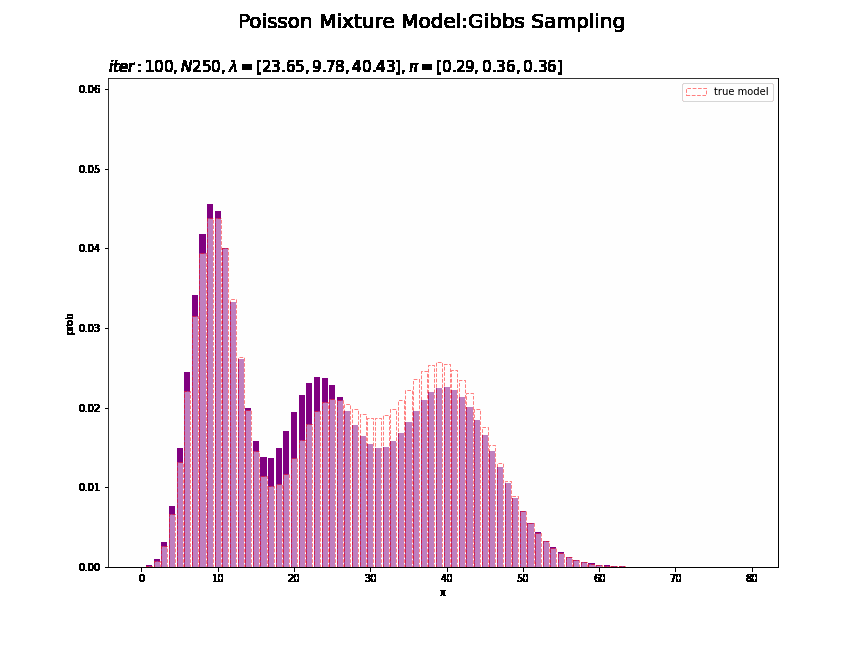
# 最後のサンプルによる混合分布を計算 res_prob = 0.0 for k in range(K): # クラスタkの分布の確率を計算 tmp_prob = poisson.pmf(k=x_line, mu=lambda_k[k]) # K個の分布の加重平均を計算 res_prob += tmp_prob * pi_k[k] # 最後のサンプルによる分布を作図 plt.figure(figsize=(12, 9)) plt.bar(x=x_line, height=res_prob, color='purple') # 最後のサンプルによる分布 plt.bar(x=x_line, height=model_prob, label='ture model', color='white', alpha=0.5, edgecolor='red', linestyle='--') # 真の分布 plt.xlabel('x') plt.ylabel('prob') plt.suptitle('Poisson Mixture Model:Gibbs Sampling', size = 20) plt.title('$iter:' + str(MaxIter) + ', N=' + str(N) + ', \lambda=[' + ', '.join([str(lmd) for lmd in np.round(lambda_k, 2)]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in np.round(pi_k, 2)]) + ']$', loc='left') plt.legend() plt.show()

観測モデルのときと同様にして作図できます。
潜在変数s_nkの最後のサンプルを使って、クラスタのヒストグラムを作成します。
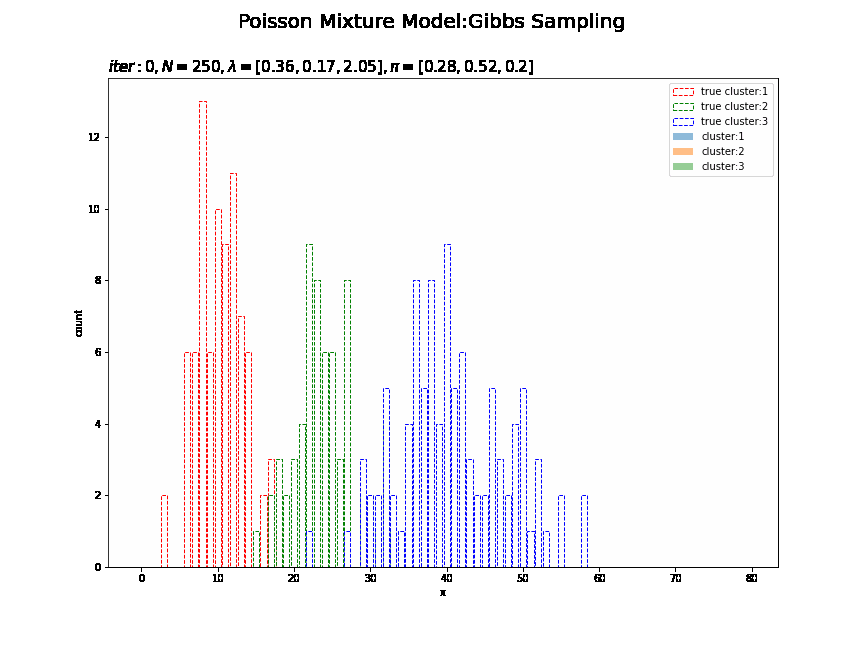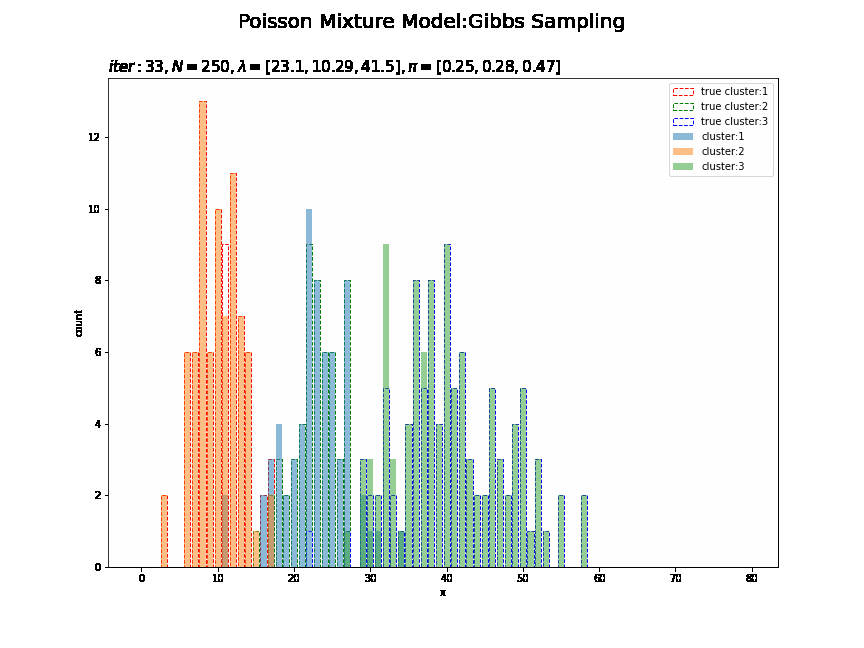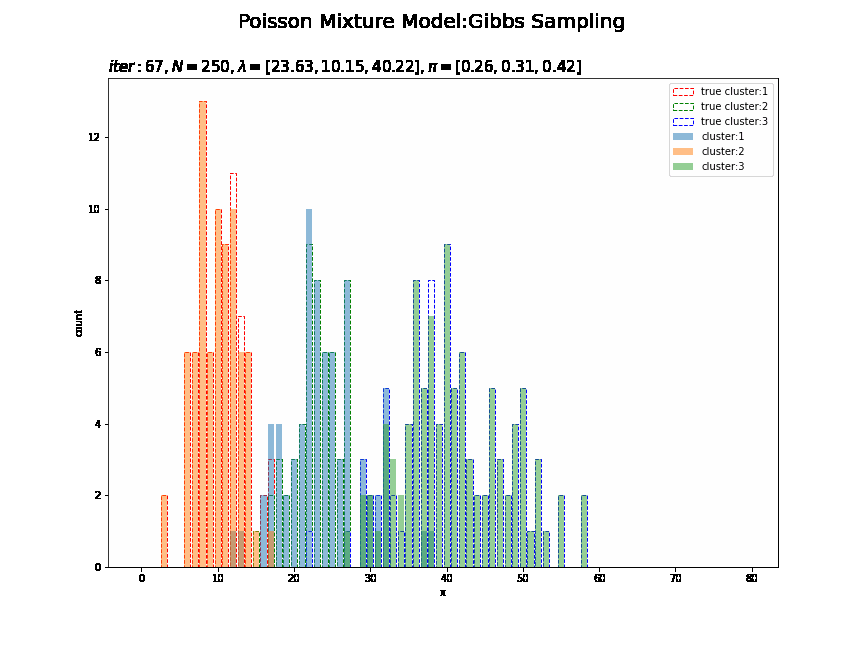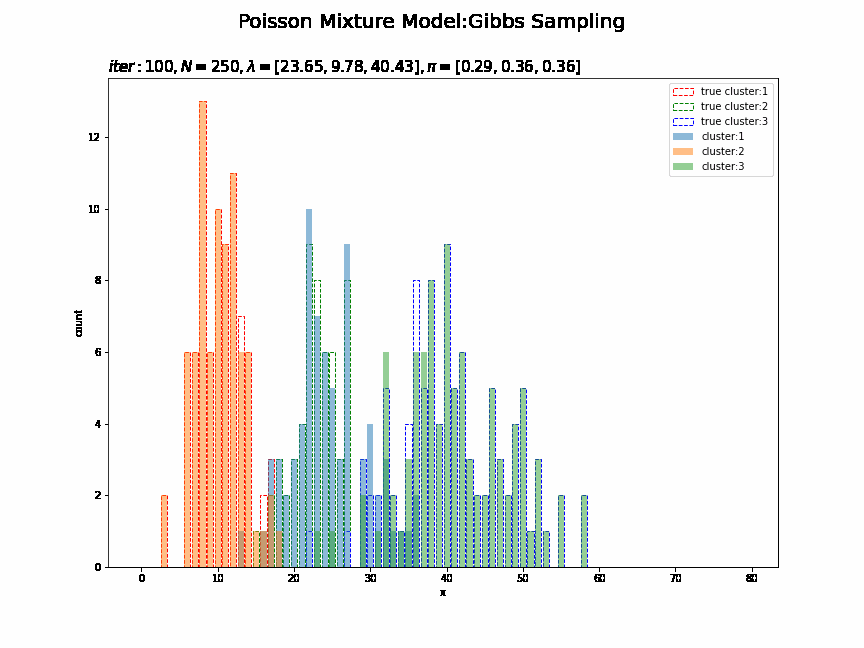
# K個の色を指定 color_list = ['red', 'green', 'blue'] # 最後のクラスタのヒストグラムを作成 plt.figure(figsize=(12, 9)) for k in range(K): plt.bar(x=x_line, height=[np.sum(x_n[s_truth_n == k] == x) for x in x_line], color='white', alpha=1, edgecolor=color_list[k], linestyle='--', label='true cluster:' + str(k + 1)) # 真のクラスタ for k in range(K): plt.bar(x=x_line, height=[np.sum(x_n[s_n == k] == x) for x in x_line], alpha=0.5, label='cluster:' + str(k + 1)) # 最後のクラスタ plt.xlabel('x') plt.ylabel('count') plt.suptitle('Poisson Mixture Model', fontsize=20) plt.title('$N=' + str(N) + ', \lambda=[' + ', '.join([str(lmd) for lmd in np.round(lambda_k, 2)]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in np.round(pi_k, 2)]) + ']$', loc='left') plt.legend() plt.show()

観測データのときと同様にして作図できます。
クラスタはランダムに決まるため、クラスタ番号は一致しません。
・超パラメータの推移の確認
超パラメータの更新値の推移を確認します。
・コード(クリックで展開)
各パラメータの学習ごとの値を格納したリストtrace_***をnp.array()でNumPy配列に変換して作図します。
# aの推移を作図 plt.figure(figsize=(12, 9)) for k in range(K): plt.plot(np.arange(MaxIter + 1), np.array(trace_a_ik).T[k], label='cluster:' + str(k + 1)) plt.xlabel('iteration') plt.ylabel('value') plt.suptitle('Gibbs Sampling', fontsize=20) plt.title('$\hat{\mathbf{a}}$', loc='left') plt.legend() plt.grid() plt.show()
# bの推移を作図 plt.figure(figsize=(12, 9)) for k in range(K): plt.plot(np.arange(MaxIter + 1), np.array(trace_b_ik).T[k], label='cluster:' + str(k + 1)) plt.xlabel('iteration') plt.ylabel('value') plt.suptitle('Gibbs Sampling', fontsize=20) plt.title('$\hat{\mathbf{b}}$', loc='left') plt.legend() plt.grid() plt.show()
# alphaの推移を作図 plt.figure(figsize=(12, 9)) for k in range(K): plt.plot(np.arange(MaxIter + 1), np.array(trace_alpha_ik).T[k], label='cluster:' + str(k + 1)) plt.xlabel('iteration') plt.ylabel('value') plt.suptitle('Gibbs Sampling', fontsize=20) plt.title('$\hat{\\bf{\\alpha}}$', loc='left') plt.legend() plt.grid() plt.show()

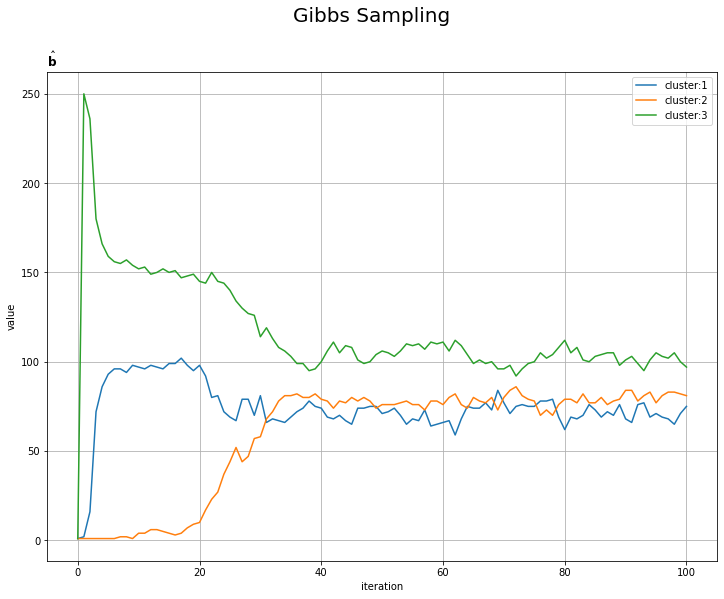
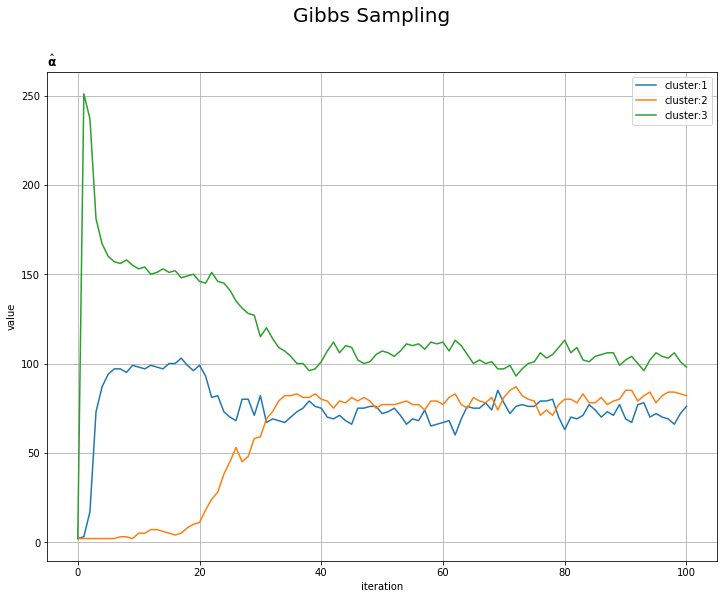
・パラメータの推移の確認
パラメータのサンプルの推移を確認します。
・コード(クリックで展開)
同様に作図します。それぞれ真の値***_truthを水平線plt.hlines()で描画しておきます。
# lambdaの推移を作図 plt.figure(figsize=(12, 9)) for k in range(K): plt.plot(np.arange(MaxIter + 1), np.array(trace_lambda_ik).T[k], label='cluster:' + str(k + 1)) plt.hlines(y=lambda_truth_k, xmin=0.0, xmax=MaxIter, label='true val', color='red', linestyle='--') # 真の値 plt.xlabel('iteration') plt.ylabel('value') plt.suptitle('Gibbs Sampling', fontsize=20) plt.title('$\hat{\\bf{\lambda}}$', loc='left') plt.legend() plt.grid() plt.show()
# piの推移を作図 plt.figure(figsize=(12, 9)) for k in range(K): plt.plot(np.arange(MaxIter + 1), np.array(trace_pi_ik).T[k], label='cluster:' + str(k + 1)) plt.hlines(y=pi_truth_k, xmin=0.0, xmax=MaxIter, label='true val', color='red', linestyle='--') # 真の値 plt.xlabel('iteration') plt.ylabel('value') plt.suptitle('Gibbs Sampling', fontsize=20) plt.title('$\hat{\\bf{\pi}}$', loc='left') plt.legend() plt.grid() plt.show()
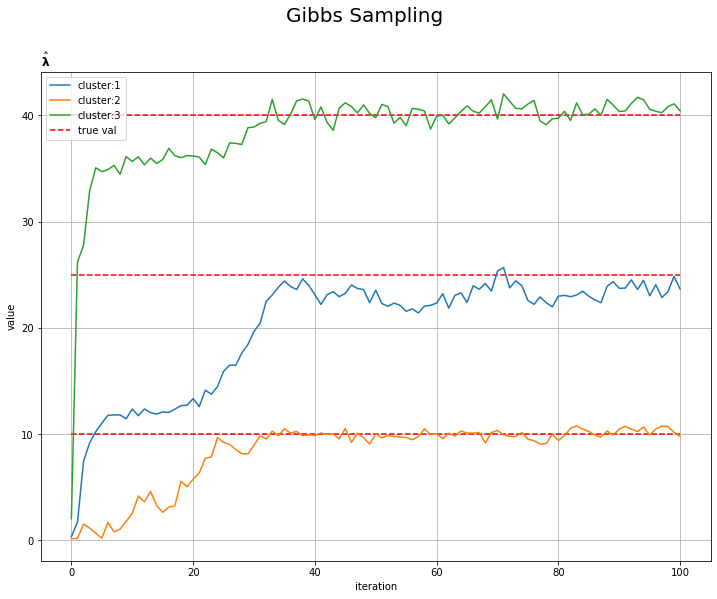

試行回数が増えるに従って真の値に近いているのを確認できます。
・おまけ:アニメーションによる推移の確認
animationモジュールを利用して、各分布の推移のアニメーション(gif画像)を作成するためのコードです。
・コード(クリックで展開)
# 追加ライブラリ import matplotlib.animation as animation
・パラメータの事後分布の推移
# 画像サイズを指定 fig = plt.figure(figsize=(12, 9)) # 作図処理を関数として定義 def update_posterior(i): # 前フレームのグラフを初期化 plt.cla() # i回目のlambdaの事後分布を計算 posterior_lambda_k = np.empty((K, len(lambda_line))) for k in range(K): posterior_lambda_k[k] = gamma.pdf(x=lambda_line, a=trace_a_ik[i][k], scale=1 / trace_b_ik[i][k]) # i回目のlambdaの事後分布を作図 for k in range(K): plt.plot(lambda_line, posterior_lambda_k[k], label='cluster:' + str(k + 1)) # 事後分布 plt.vlines(x=lambda_truth_k[k], ymin=0.0, ymax=np.max(posterior_lambda_k), label='true val', color='red', linestyle='--') # 真の値 plt.xlabel('$\lambda$') plt.ylabel('density') plt.suptitle('Gamma Distribution:Gibbs Sampling', fontsize=20) plt.title('$iter:' + str(i) + ', N=' + str(N) + ', \hat{a}=[' + ', '.join([str(a) for a in trace_a_ik[i]]) + ']' + ', \hat{b}=[' + ', '.join([str(b) for b in trace_b_ik[i]]) + ']$', loc='left', fontsize=15) plt.legend() # gif画像を作成 posterior_anime = animation.FuncAnimation(fig, update_posterior, frames=MaxIter + 1, interval=100) posterior_anime.save("ch4_3_2_Posterior.gif")
・サンプルしたパラメータによる分布の推移
・コード(クリックで展開)
# 分布の最大値を計算 max_prob = np.max(poisson.pmf(k=x_line, mu=np.max(trace_lambda_ik))) # 画像サイズを指定 fig = plt.figure(figsize=(12, 9)) # 作図処理を関数として定義 def update_model(i): # 前フレームのグラフを初期化 plt.cla() # i回目のサンプルによる混合分布を計算 res_prob = 0.0 for k in range(K): # クラスタkの分布の確率を計算 tmp_prob = poisson.pmf(k=x_line, mu=trace_lambda_ik[i][k]) # K個の分布の加重平均を計算 res_prob += tmp_prob * trace_pi_ik[i][k] # i回目のサンプルによる分布を作図 plt.bar(x=x_line, height=res_prob, color='purple') # サンプルによる分布 plt.bar(x=x_line, height=model_prob, label='true model', color='white', alpha=0.5, edgecolor='red', linestyle='--') # 真の分布 plt.xlabel('x') plt.ylabel('prob') plt.suptitle('Poisson Mixture Model:Gibbs Sampling', size = 20) plt.title('$iter:' + str(i) + ', N' + str(N) + ', \lambda=[' + ', '.join([str(lmd) for lmd in np.round(trace_lambda_ik[i], 2)]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in np.round(trace_pi_ik[i], 2)]) + ']$', loc='left', fontsize=15) plt.ylim(0.0, max_prob) plt.legend() # gif画像を作成 model_anime = animation.FuncAnimation(fig, update_model, frames=MaxIter + 1, interval=100) model_anime.save("ch4_3_2_Model.gif")
・クラスタのヒストグラムの推移
# K個の色を指定 color_list = ['red', 'green', 'blue'] # 作図用のxの点を作成 x_line = np.arange(0, 2 * np.max(lambda_truth_k)) # 画像サイズを指定 fig = plt.figure(figsize=(12, 9)) # 作図処理を関数として定義 def update_cluster(i): # 前フレームのグラフを初期化 plt.cla() # i回目のクラスタの散布図を作成 for k in range(K): plt.bar(x=x_line, height=[np.sum(x_n[s_truth_n == k] == x) for x in x_line], color='white', alpha=1, edgecolor=color_list[k], linestyle='--', label='true cluster:' + str(k + 1)) # 真のクラスタ for k in range(K): plt.bar(x=x_line, height=[np.sum(x_n[np.array(trace_s_in[i]) == k] == x) for x in x_line], alpha=0.5, label='cluster:' + str(k + 1)) # サンプルしたクラスタ plt.xlabel('x') plt.ylabel('count') plt.suptitle('Poisson Mixture Model:Gibbs Sampling', fontsize=20) plt.title('$iter:' + str(i) + ', N=' + str(N) + ', \lambda=[' + ', '.join([str(lmd) for lmd in np.round(trace_lambda_ik[i], 2)]) + ']' + ', \pi=[' + ', '.join([str(pi) for pi in np.round(trace_pi_ik[i], 2)]) + ']$', loc='left', fontsize=15) plt.legend() # gif画像を作成 cluster_anime = animation.FuncAnimation(fig, update_cluster, frames=MaxIter + 1, interval=100) cluster_anime.save("ch4_3_2_Cluster.gif")
(よく理解していないので、animationの解説は省略…とりあえずこれで動きます……)
参考文献
- 須山敦志『ベイズ推論による機械学習入門』(機械学習スタートアップシリーズ)杉山将監修,講談社,2017年.
おわりに
現時点で全部実装できた。あとは解説文を書くだけ。
2021年5月7日は、モーニング娘。'21の佐藤優樹さんの22歳のお誕生日です!!!!
22歳の年が来た♪♪おめでとうございます!早くライブ行きたーい。
【次節の内容】