はじめに
『スタンフォード ベクトル・行列からはじめる最適化数学』の学習ノートです。
「数式の行間埋め」や「Pythonを使っての再現」によって理解を目指します。本と一緒に読んでください。
この記事は4.3節「k平均法」の内容です。
多次元混合ガウス分布の乱数に対するk平均法によるクラスタリングを実装します。
【前の内容】
【他の内容】
【今回の内容】
混合ガウス乱数のクラスタリング
多次元混合ガウス分布(maltivariation gaussian mixture distribution)の乱数に対して、k平均法(k-means algorithm)によりクラスタリング(clustering)を行います。
多次元混合ガウス分布については「多次元混合ガウス分布の定義式 - からっぽのしょこ」、代表値との距離については「【Python】3.2:最近傍の可視化【『スタンフォード線形代数入門』のノート】 - からっぽのしょこ」を参照してください。
利用するライブラリを読み込みます。
# 利用ライブラリ import numpy as np from scipy.stats import multivariate_normal import matplotlib.pyplot as plt from matplotlib.animation import FuncAnimation
2次元の場合
まずは、2次元空間(平面)上のベクトル(2次元のデータ)のクラスタリングを可視化します。
トイデータの作成
クラスタリングを行う2次元混合ガウス分布の乱数を生成します。
・作図コード(クリックで展開)
データの生成分布を設定します。
# 真のクラスタ数を指定 K_truth = 3 # K個の平均ベクトルを指定 mu = np.array( [[0.0, 5.0], [5.0, -10.0], [-10.0, -5.0]] ) # K個の分散共分散行列を指定 sigma = np.array( [[[36.0, 10.0], [10.0, 25.0]], [[9.0, -1.3], [-1.3, 16.0]], [[25.0, -3.2], [-3.2, 16.0]]] ) # 混合比率を指定 pi = np.array([0.45, 0.25, 0.3])
真のクラスタ数を指定します。次元数(本では
)は
とします。
個の平均ベクトル
と共分散行列
を
mu, sigmaとして値を指定します。クラスタの平均ベクトルと分散共分散行列はそれぞれ次の形状です。
は
のときの(クラスタjのデータ)
の平均、
は
の標準偏差、
は
の分散、
は
の共分散です。
は正定値行列である必要があります。
混合比率(クラスタの割り当て確率)、
、
を
piとして値を指定します。
確率密度の等高線を描画するのに使う点を作成します。データの生成自体には不要な処理です。
# 各軸の値を作成 x1_vals = np.linspace( np.min(mu[:, 0] - np.sqrt(sigma[:, 0, 0]) * 3.0), np.max(mu[:, 0] + np.sqrt(sigma[:, 0, 0]) * 3.0), num=300 ) x2_vals = np.linspace( np.min(mu[:, 1] - np.sqrt(sigma[:, 1, 1]) * 3.0), np.max(mu[:, 1] + np.sqrt(sigma[:, 1, 1]) * 3.0), num=300 ) print(x1_vals[:5].round(1)) print(x2_vals[:5].round(1)) # 作図用の点を作成 x1_grid, x2_grid = np.meshgrid(x1_vals, x2_vals) print(x1_grid[:5, :5].round(1)) print(x2_grid[:5, :5].round(1)) # 作図用の点の形状を保存 x_dim = x1_grid.shape print(x_dim) # 計算用の点を作成 x_points = np.stack([x1_grid.flatten(), x2_grid.flatten()], axis=1) print(x_points[:5].round(1)) print(x_points.shape)
[-25. -24.9 -24.7 -24.6 -24.4]
[-22. -21.9 -21.7 -21.6 -21.4]
[[-25. -24.9 -24.7 -24.6 -24.4]
[-25. -24.9 -24.7 -24.6 -24.4]
[-25. -24.9 -24.7 -24.6 -24.4]
[-25. -24.9 -24.7 -24.6 -24.4]
[-25. -24.9 -24.7 -24.6 -24.4]]
[[-22. -22. -22. -22. -22. ]
[-21.9 -21.9 -21.9 -21.9 -21.9]
[-21.7 -21.7 -21.7 -21.7 -21.7]
[-21.6 -21.6 -21.6 -21.6 -21.6]
[-21.4 -21.4 -21.4 -21.4 -21.4]]
(300, 300)
[[-25. -22. ]
[-24.9 -22. ]
[-24.7 -22. ]
[-24.6 -22. ]
[-24.4 -22. ]]
(90000, 2)
この例では軸ごとに、平均を中心に標準偏差の3倍を範囲とします。
軸ごとの値x*_valsを作成して、np.meshgrid()で格子状の点(全ての組み合わせ)x*_gridを作成します。また、x*_gridをそれぞれ列として結合して、2列の配列を作成します。
x*_gridの同じインデックスの値、x_pointsの行が確率変数の値に対応します。
混合ガウス分布の確率密度を計算します。
# 確率密度を計算 dens = np.sum( [pi[j] * multivariate_normal.pdf(x=x_points, mean=mu[j], cov=sigma[j]) for j in range(K_truth)], axis=0 ) print(dens[:5]) print(dens.shape)
[3.28052487e-10 3.63080417e-10 4.01620357e-10 4.43994078e-10
4.90549144e-10]
(90000,)
リスト内包表記のfor文を使って、クラスタごとに確率密度を計算し、混合比率を用いて加重平均を計算します。
各クラスタの(混合ではない)多次元ガウス分布の確率密度は、SciPyライブラリのmultivariate_normalモジュールの確率密度関数pdf()で計算できます。確率変数の引数xに計算用の点x_points、平均ベクトルの引数meanに各クラスタの平均ベクトルmu[j]、分散共分散行列の引数covに各クラスタの分散共分散行列sugma[j]を指定します。
各データに真のクラスタを割り当てます。
# データ数を指定 N = 100 # 真のクラスタを生成 c_truth_onehot = np.random.multinomial(n=1, pvals=pi, size=N) print(c_truth_onehot[:5]) print(c_truth_onehot.shape) # 真のクラスタ番号を抽出 _, c_truth = np.where(c_truth_onehot == 1) print(c_truth[:5]) print(c_truth.shape) # クラスタごとのデータ数を集計 G_num = np.sum(c_truth_onehot, axis=0) print(G_num) print(G_num.shape)
[[0 1 0]
[0 0 1]
[0 0 1]
[1 0 0]
[0 0 1]]
(100, 3)
[1 2 2 0 2]
(100,)
[47 23 30]
(3,)
データ数(ベクトル数)を指定します。
多項分布の乱数生成関数np.random.multinomial()の試行回数の引数nに1を指定して、カテゴリ分布の乱数を生成します。パラメータ(割り当て確率)の引数pvalsに混合比率pi、サンプルサイズの引数sizeにデータ数Nを指定します。
one-hotベクトル(クラスタ番号に対応する列が1でそれ以外の列が0の行)をまとめたN行K_truth列の配列が出力されるので、np.where()で行ごとに値が1の列番号を抽出して、データごとの真のクラスタ番号、
とします。
各列の値が1の行数が、各クラスタが割り当てられたデータ数です。他の要素は0なので、列ごとの和で得られます。
データ(ベクトル)を生成します。
# データを生成 X = np.array( [np.random.multivariate_normal(mean=mu[j], cov=sigma[j], size=1).flatten() for j in c_truth] ) print(X[:5]) print(X.shape)
[[ 4.76456936 -8.15120039]
[-15.75284292 -3.86563593]
[ -9.33917747 -6.14028522]
[ 7.06122445 6.89315561]
[ -8.39557913 -10.8988496 ]]
(100, 2)
クラス内包表記のfor文を使って、c_truthから順番にクラスタ番号を取り出して、対応するクラスタの多次元ガウス分布の乱数をnp.random.multivariate_normal()で生成します。平均ベクトルの引数meanに各クラスタの平均ベクトルmu[j]、分散共分散行列の引数covに各クラスタの分散共分散行列sigma[j]、サンプルサイズの引数sizeに1を指定します。
N行2列の配列が出力されるので、データ、
とします。
生成分布の等高線図とデータの散布図を作成します。
# 生成分布とデータを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white') cnf = ax.contour(x1_grid, x2_grid, dens.reshape(x_dim), linestyles='--', zorder=0) # 生成分布 for j in range(K_truth): G_j, = np.where(c_truth == j) # クラスタjのデータインデック ax.scatter(x=X[G_j, 0], y=X[G_j, 1], s=50, label=str(j+1), zorder=1) # サンプルデータ ax.set_title('N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + ')', loc='left') fig.suptitle('Gaussian mixture distribution', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.grid() ax.legend(title='cluster') fig.colorbar(cnf, label='density') ax.set_aspect('equal') plt.show()
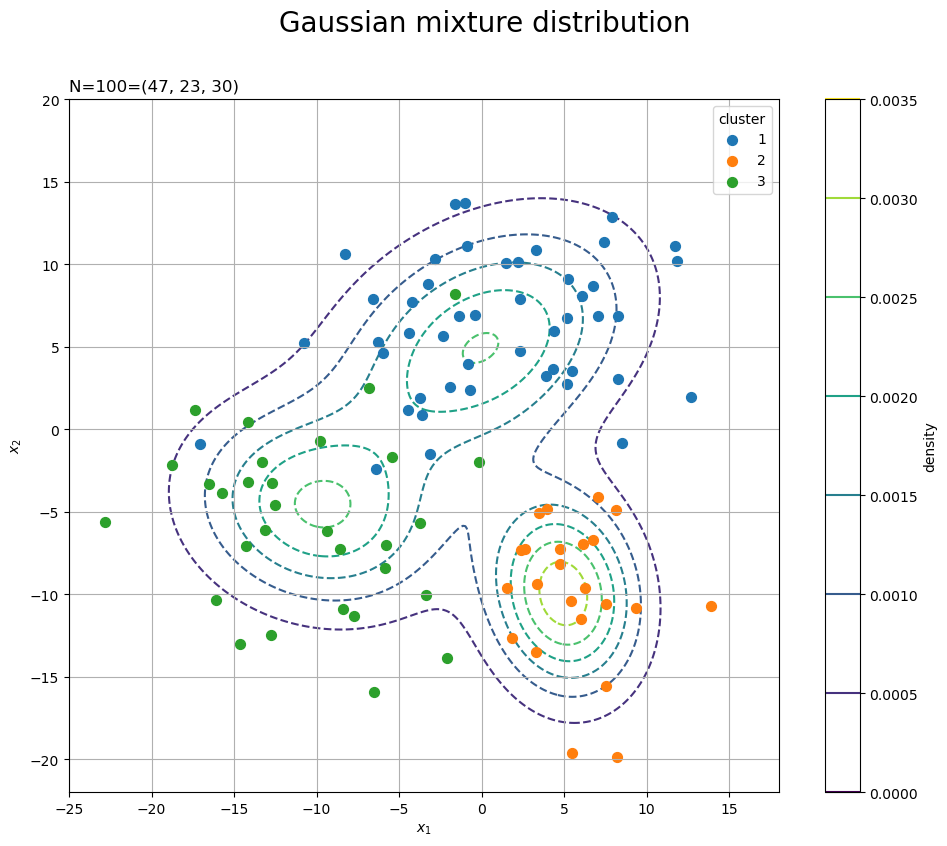
混合多次元ガウス分布の確率密度の等高線をaxes.contour()で描画します。
また、クラスタごとに多次元ガウス分布の乱数の散布図をaxes.scatter()で描画します。c_truthから値がjのインデックスを抽出してG_jとします。さらに、XからG_jに含まれる行を取り出して、色分けして描画します。
目安として、目的関数を計算します。
# 目的関数(ノルムの2乗平均)を計算 J_truth = np.array( [np.sum(np.linalg.norm(X[c_truth == j] - mu[j], axis=1)**2) / N for j in range(K_truth)] ) print(J_truth)
[27.01011683 5.88060014 17.64819944]
生成分布の平均ベクトルを代表値として、「クラスタリング」で確認する目的関数と同様に、次の式を計算します。
個の
を
J_truthとしておき、作図時にを計算します。
は真のクラスタ
の集合
を表します。
は、ユークリッドノルムで、
np.linalg.norm()で計算できます。
真のクラスタをグラフで確認します。
# カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(K_truth) / K_truth) # 2Dベクトルの真のクラスタを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white') for j in range(K_truth): G_j, = np.where(c_truth == j) # クラスタjのデータのインデック ax.plot([mu[j, 0].repeat(len(G_j)), X[G_j, 0]], [mu[j, 1].repeat(len(G_j)), X[G_j, 1]], color=colors[j], linewidth=1, linestyle=':', zorder=0) # 対応線 ax.scatter(x=mu[j, 0], y=mu[j, 1], edgecolor=colors[j], facecolor='white', marker='s', s=100, zorder=1) # 平均値 ax.scatter(x=X[G_j, 0], y=X[G_j, 1], color=colors[j], s=20, label=str(j+1), zorder=2) # サンプルデータ ax.set_title('N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + '), ' + 'J=' + str(np.sum(J_truth).round(1)) + '=(' + ', '.join(map(str, J_truth.round(1))) + ')', loc='left') fig.suptitle('true cluster', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.grid() ax.legend(title='cluster') ax.set_aspect('equal') plt.show()
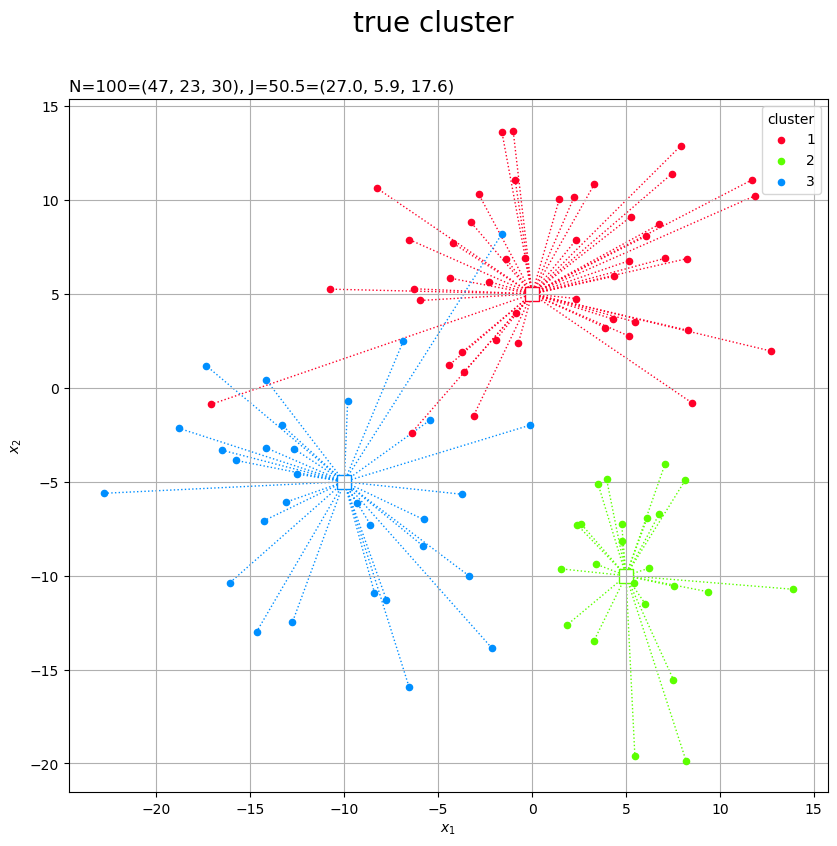
各データを塗りつぶしの丸、真の平均値を白抜きの四角として描画します。
クラスタごとに色分けするために、plt.get_cmap()にカラーマップ名を指定して、クラスタごとに色付けします。
以上で、クラスタリングに利用する人工データを作成できました。
クラスタリング
2次元ベクトルに対するk平均法を実装します。
2通りの方法で初期値を設定できます。
1つ目は、クラスタの初期値をランダムに割り当てて、クラスタごとに代表値として平均値を計算します。
# クラスタ数の初期値を指定 K = 10 # ランダムにクラスタを割り当て c_onehot = np.random.multinomial(n=1, pvals=np.repeat(1, K)/K, size=N) print(c_onehot[:5]) # クラスタ番号を抽出 _, c = np.where(c_onehot == 1) print(c[:5]) # クラスタごとのデータ数を集計 G_num = np.sum(c_onehot, axis=0) print(G_num) # クラスタの代表値(平均値)を計算 Z = np.array( [np.mean(X[c == j], axis=0) for j in range(K)] ) print(Z[:5]) # 目的関数(ノルムの2乗平均)を計算 J = np.array( [np.sum(np.linalg.norm(X[c == j] - Z[j], axis=1)**2) / N for j in range(K)] ) print(J[:5])
[[0 0 1 0 0 0 0 0 0 0]
[1 0 0 0 0 0 0 0 0 0]
[0 0 0 0 0 0 0 1 0 0]
[0 0 1 0 0 0 0 0 0 0]
[1 0 0 0 0 0 0 0 0 0]]
[2 0 7 2 0]
[11 12 15 11 5 13 10 6 7 10]
[[-2.11756701 -1.17656645]
[ 1.45066723 2.03206318]
[ 1.25141833 -2.75095687]
[-0.49235447 -4.05065165]
[-2.37184145 4.71364666]]
[10.97033869 18.36041448 17.14423579 14.55457911 3.09226981]
本来は真のクラスタ数は分からないものなので、クラスタ数
の初期値を指定します。
データの生成時と同様にして、データごとのクラスタ、
の初期値を割り当てます。
各クラスタが割り当てられたデータの平均値を代表値、
とします。
は(初期値や推定値の)クラスタ
の集合
、
は
の要素数を表します。
各クラスタが割り当てられた要素数(データ数)は、データの生成時と同様にして、G_numとします。
各データと割り当てられたクラスタの代表値
の距離
の2乗平均を目的関数とします。
個の
を
Jとしておき、作図時にを計算します。
真のクラスタのときと同様にして、クラスタの初期値をグラフで確認します。
# カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(K) / K) # 2Dベクトルのクラスタを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white') for j in range(K): G_j, = np.where(c == j) # クラスタjのデータのインデック ax.plot([Z[j, 0].repeat(len(G_j)), X[G_j, 0]], [Z[j, 1].repeat(len(G_j)), X[G_j, 1]], color=colors[j], linewidth=1, linestyle=':', zorder=0) # 対応線 ax.scatter(x=Z[j, 0], y=Z[j, 1], edgecolor=colors[j], facecolor='white', marker='s', s=100) # 代表値 ax.scatter(x=X[G_j, 0], y=X[G_j, 1], color=colors[j], s=20, label=str(j+1)) # サンプルデータ ax.set_title('iter:0, ' + 'N=' + str(N) + '=(' + ', '.join(str(val) for val in G_num) + '), ' + 'J=' + str(np.sum(J).round(1)) + '=(' + ', '.join(map(str, J.round(1))) + ')', loc='left') fig.suptitle('initial cluster', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.grid() ax.legend(title='cluster') ax.set_aspect('equal') plt.show()
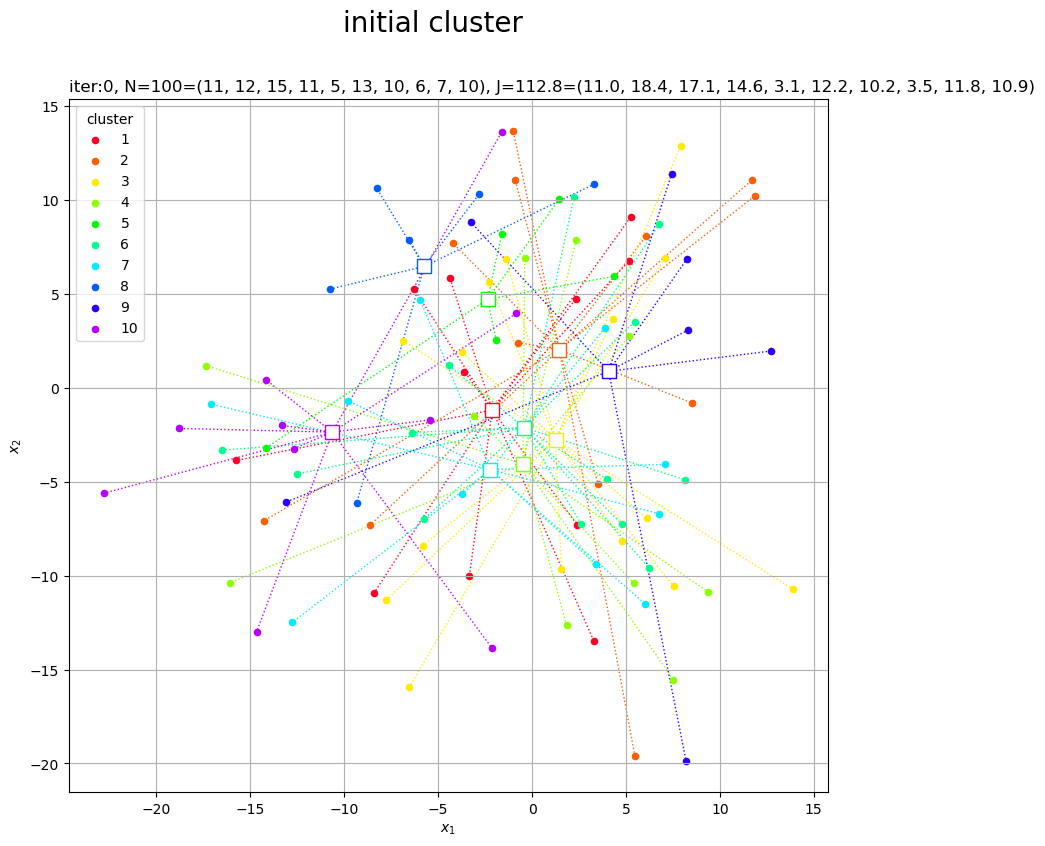
ランダムに割り当てられたデータの平均値なので、代表値がデータ全体の中心付近に集まっています。
2つ目は、代表値をランダムに設定して、データごとに代表値との距離が近い(ノルムが小さい)クラスタを割り当てます。
# クラスタ数の初期値を指定 K = 10 # ランダムにクラスタの代表値を設定 Z = np.array( [np.random.uniform(low=X[:, 0].min(), high=X[:, 0].max(), size=K), np.random.uniform(low=X[:, 1].min(), high=X[:, 1].max(), size=K)] ).T print(Z[:5]) # ノルムが最小のクラスタを割り当て c = np.argmin( [np.linalg.norm(X - Z[j], axis=1) for j in range(K)], axis=0 ) print(c[:5]) # クラスタごとのデータ数を集計 G_num = np.array( [np.sum(c == j) for j in range(K)] ) print(G_num) # 目的関数(ノルムの2乗平均)を計算 J = np.array( [np.sum(np.linalg.norm(X[c == j] - Z[j], axis=1)**2) / N for j in range(K)] ) print(J[:5])
[[-12.86969694 10.39565196]
[ 9.29229126 13.58730506]
[ 3.62484662 4.05864789]
[-15.96730452 -0.04264619]
[ -6.55348377 4.85129958]]
[7 3 3 2 5]
[ 1 7 19 15 20 14 0 17 4 3]
[0.21124562 2.20122433 3.79361867 4.47382537 5.44145877]
クラスタ数の初期値を指定して、
個の代表値
の初期値をランダムに生成します。この例では、次元ごとに
の最小値から最大値を範囲として、
np.random.uniform()で一様乱数を作成します。
各データと各クラスタの代表値
との距離
が最小となるクラスタ
をそのデータのクラスタ
とします。
最小値のインデックスはnp.argmin()で得られます。
こちらの方法では、one-hotベクトルのクラスタ番号が不要なので、cに含まれる1からKの要素数をカウントして、クラスタごとのデータ数G_numとします。
1つ目の方法と同じコードで、個の
を
Jとして計算します。
1つ目の方法と同じコードで、クラスタの初期値をグラフで確認します。
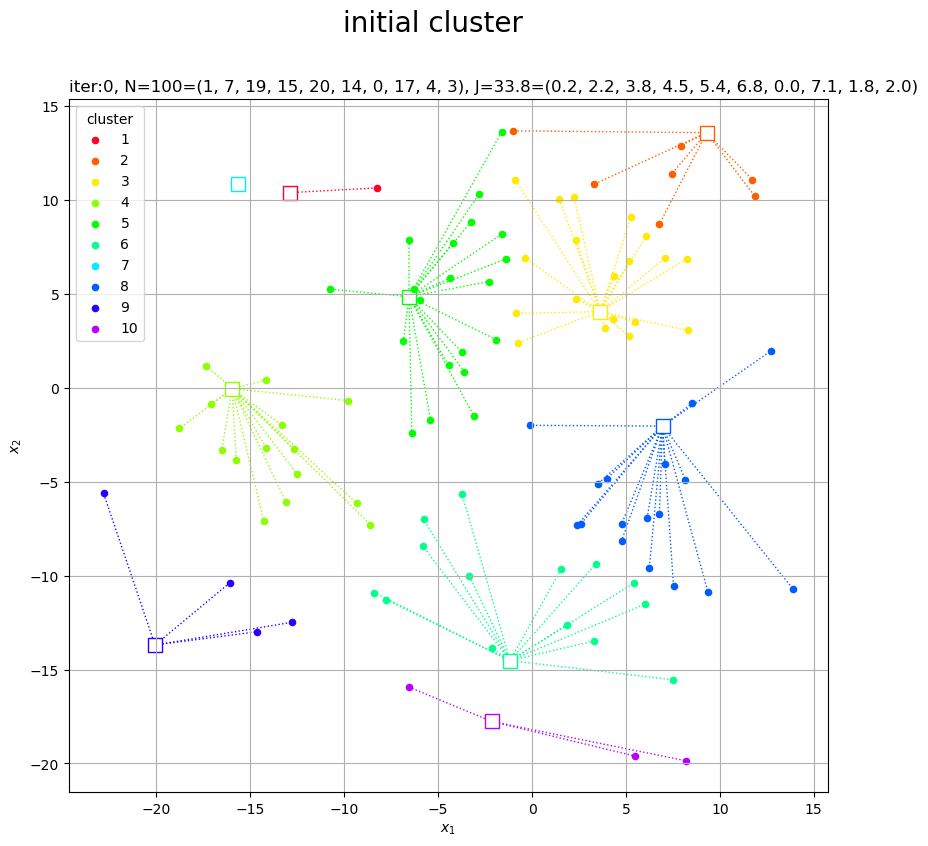
代表値がばらけるので、ある程度まとまったクラスタができています。データが割り当てられないクラスタができることがあります。
k平均法によりクラスタと代表値を繰り返し更新します。
# クラスタの最低割り当て数を指定 G_num_lower = 10 # 更新量の閾値を指定 threshold = 0.001 # 初期値を記録 trace_Z = [Z] trace_c = [c] trace_G = [G_num] trace_J = [J] # 繰り返し試行 cnt = 0 old_J_sum = np.sum(J) while True: # 試行回数をカウント cnt += 1 print('--- iter:' + str(cnt) + ' ---') # クラスタの代表値(平均値)を計算 Z = np.array( [np.mean(X[c == j], axis=0) for j in range(K) if G_num[j] >= G_num_lower] ) # クラスタ数を再設定 K = len(Z) print('K=' + str(K)) # ノルムが最小のクラスタを割り当て c = np.argmin( [np.linalg.norm(X - Z[j], axis=1) for j in range(K)], axis=0 ) # クラスタごとのデータ数を集計 G_num = np.array( [np.sum(c == j) for j in range(K)] ) print('|G|=' + str(G_num)) # 目的関数(ノルムの2乗平均)を計算 J = np.array( [np.sum(np.linalg.norm(X[c == j] - Z[j], axis=1)**2) / N for j in range(K)] ) J_sum = np.sum(J) print('J=' + str(J_sum.round(5))) # 更新値を記録 trace_Z.append(Z) trace_c.append(c) trace_G.append(G_num) trace_J.append(J) # 更新量が閾値未満なら終了 if abs(J_sum - old_J_sum) < threshold: break # 目的関数の値を保存 old_J_sum = J_sum
--- iter:1 ---
K=5
|G|=[26 19 22 14 19]
J=28.81495
--- iter:2 ---
K=5
|G|=[25 19 24 13 19]
J=25.93017
--- iter:3 ---
K=5
|G|=[25 19 24 12 20]
J=25.6878
--- iter:4 ---
K=5
|G|=[25 19 24 10 22]
J=25.34453
--- iter:5 ---
K=5
|G|=[25 18 24 9 24]
J=24.64732
--- iter:6 ---
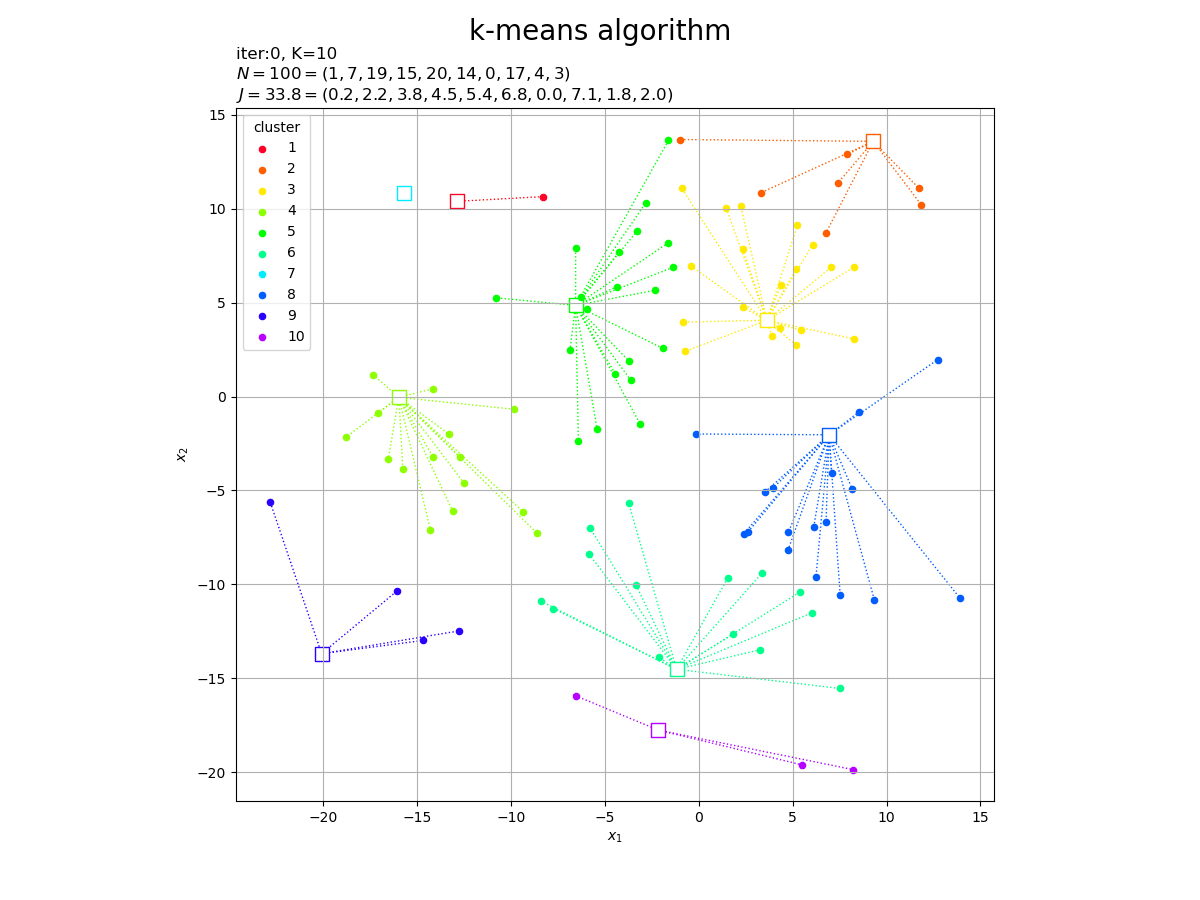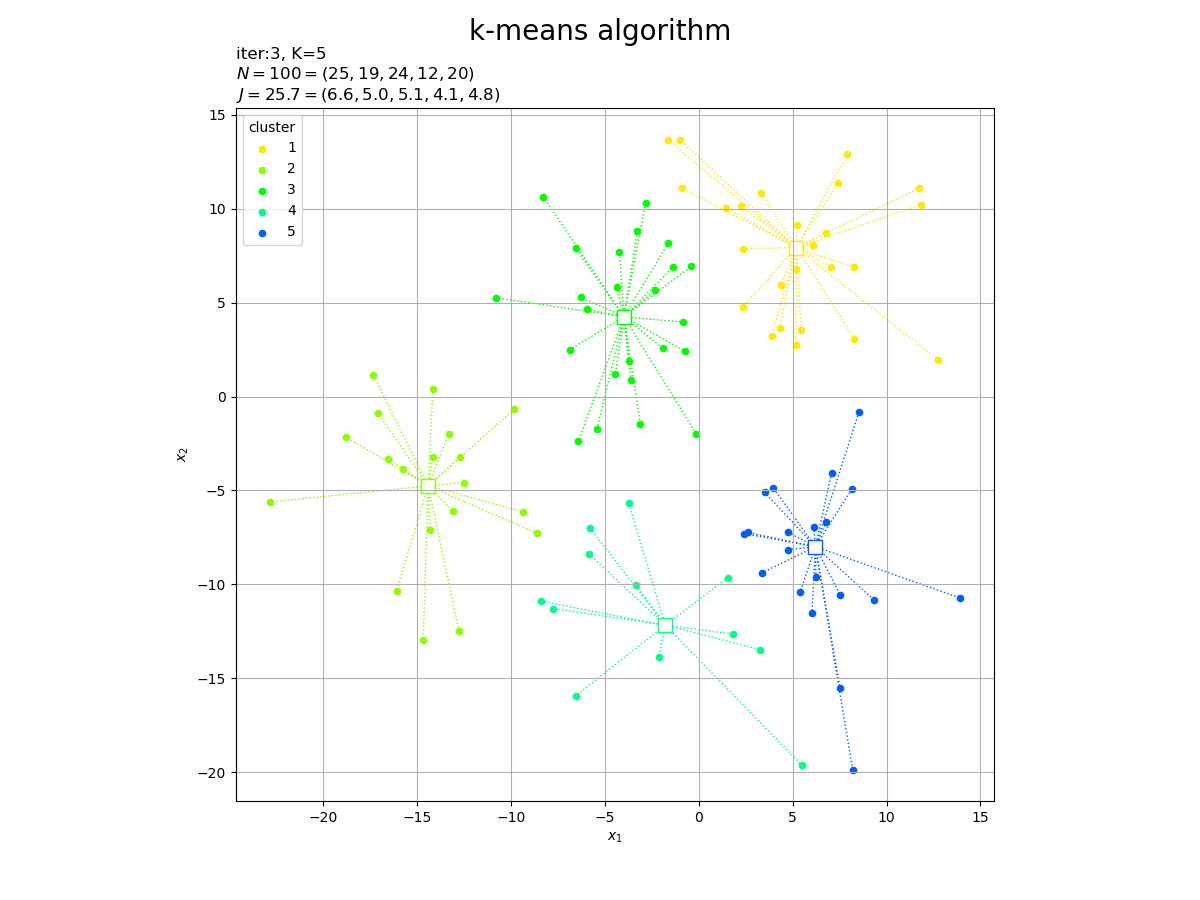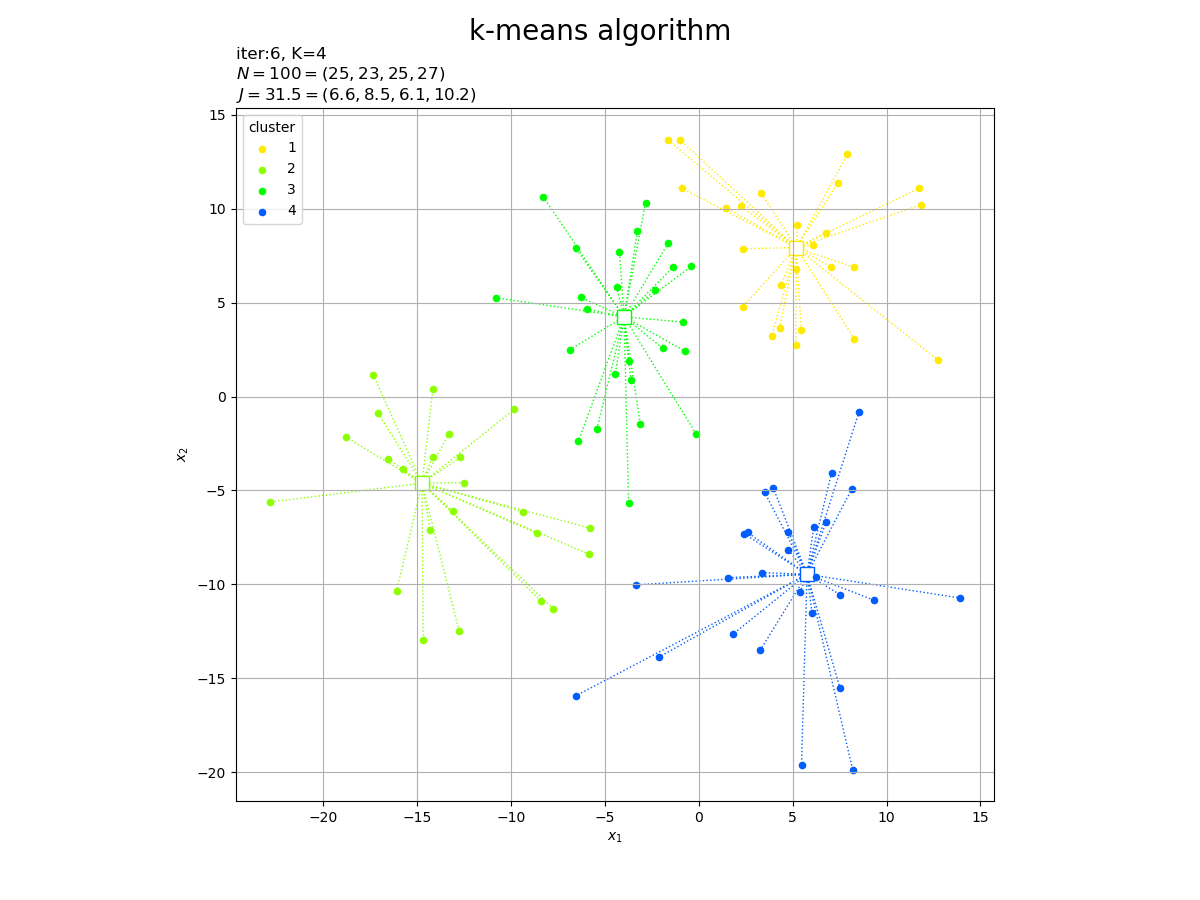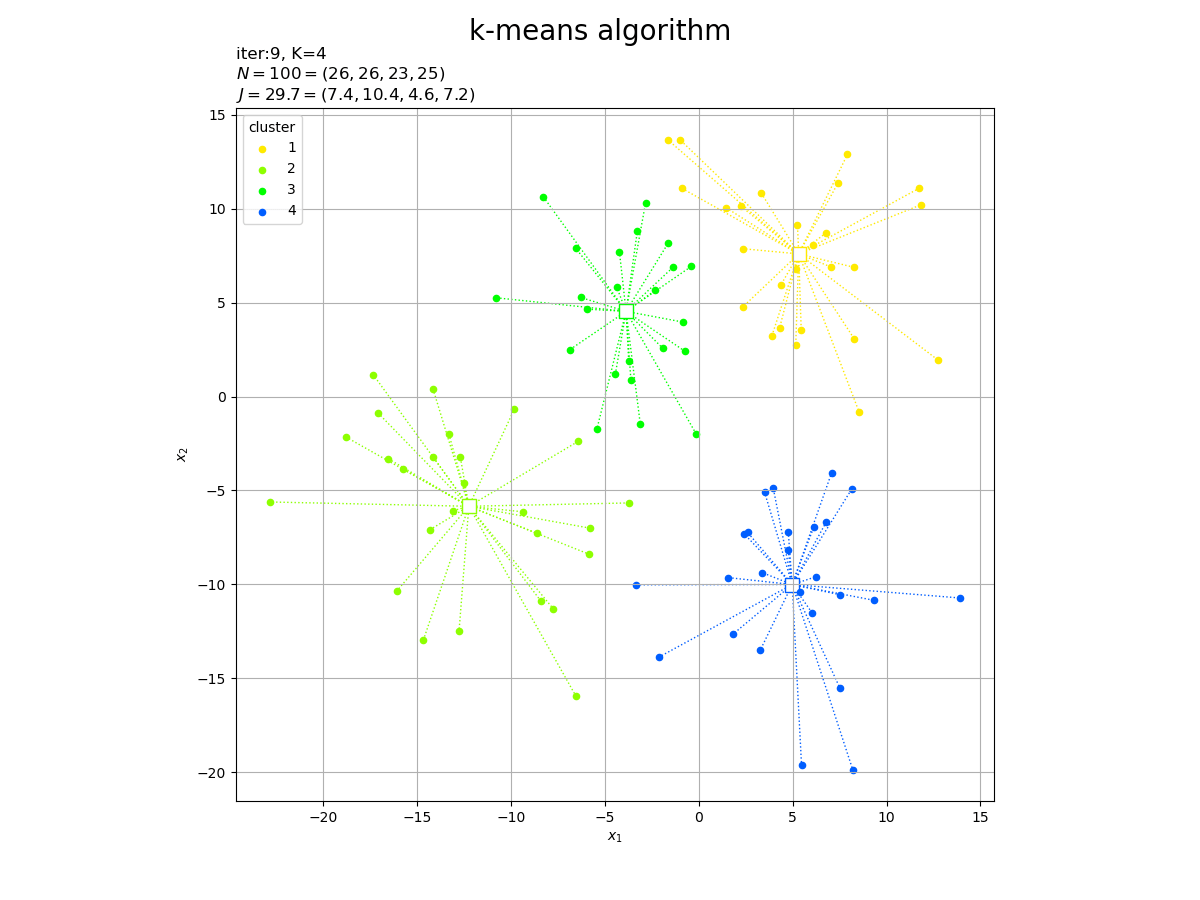
K=4
|G|=[25 23 25 27]
J=31.48424
--- iter:7 ---
K=4
|G|=[26 25 24 25]
J=30.02559
--- iter:8 ---
K=4
|G|=[26 26 23 25]
J=29.78286
--- iter:9 ---
K=4
|G|=[26 26 23 25]
J=29.74205
--- iter:10 ---
K=4
|G|=[26 26 23 25]
J=29.74205
クラスタに含まれるデータ数が一定数未満になると、そのクラスタを削除することにします。
下限値をG_num_lowerとして指定して、クラスタjのデータ数G_num[j]がG_num_lower以上のときのみ代表値(平均値)を計算します。クラスタが削除される(計算されないクラスタがある)と、クラスタ番号(行インデックス)が変わります。
Zの行数をKとして、クラスタ数を更新します。
クラスタの割り当てや、目的関数の計算については、これまでと同じです。
閾値thresholdを指定し、前ステップの値をold_J_sumとしておき、更新値J_sumとの差の絶対値が閾値未満になると、収束したとみなしてbreakでループ処理を終了します。
更新推移の確認用に、値(配列)をtrace_*に記録していきます。
クラスタの更新値(収束結果)をグラフで確認します。
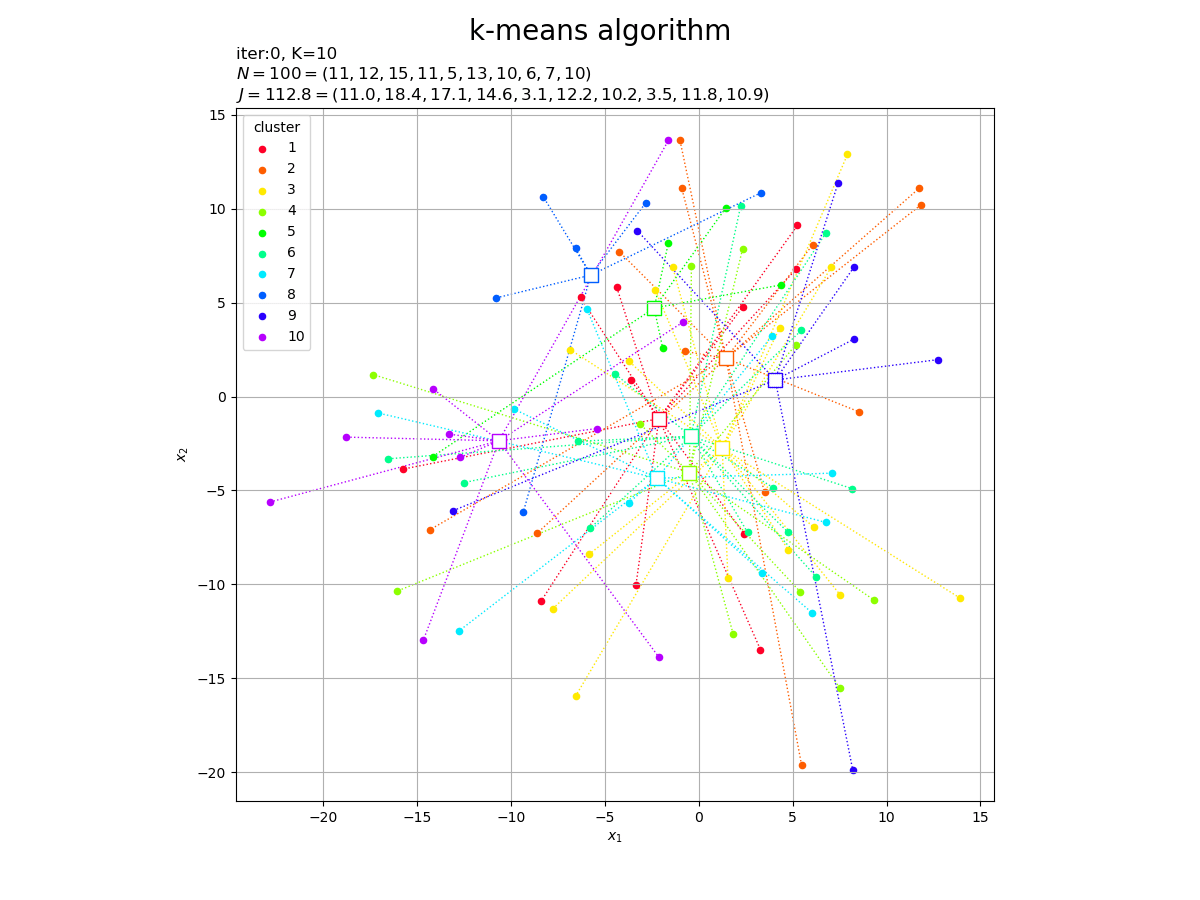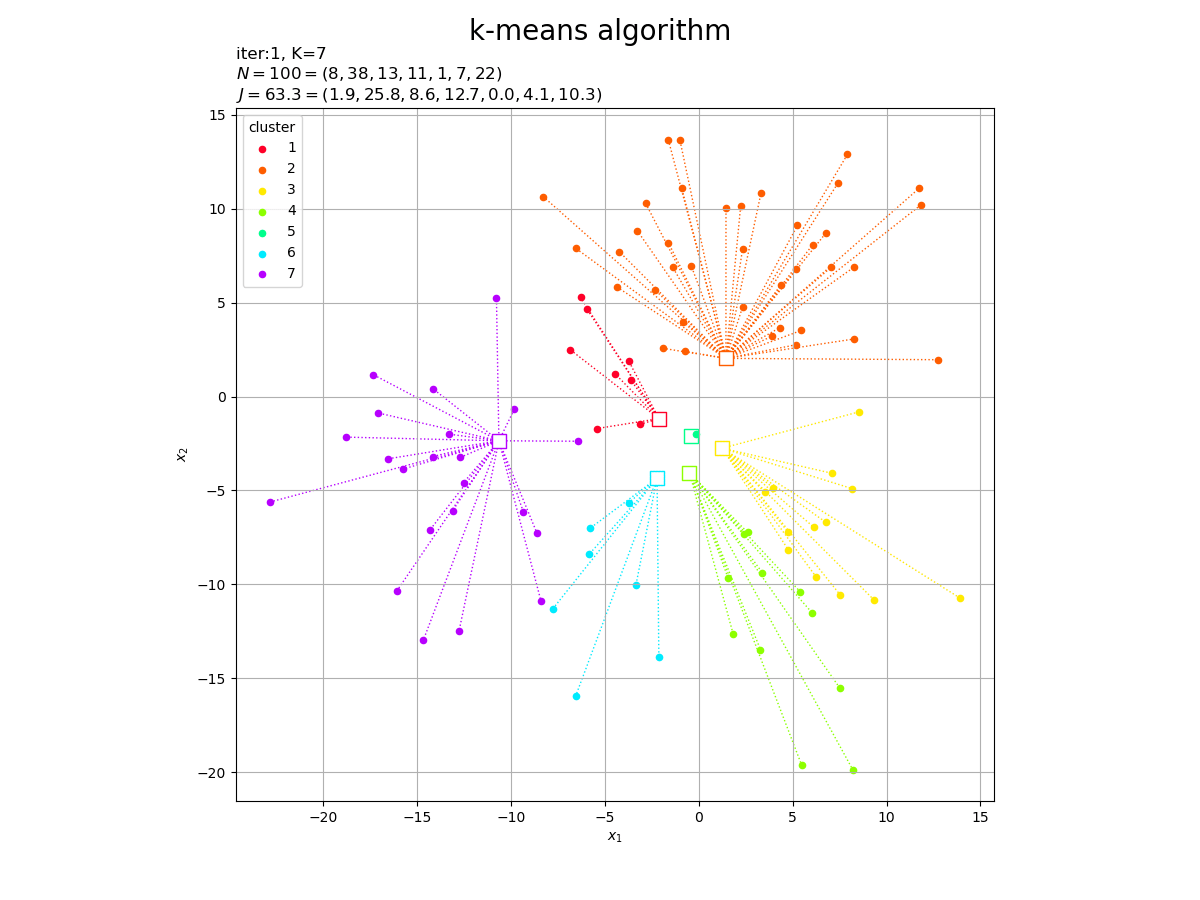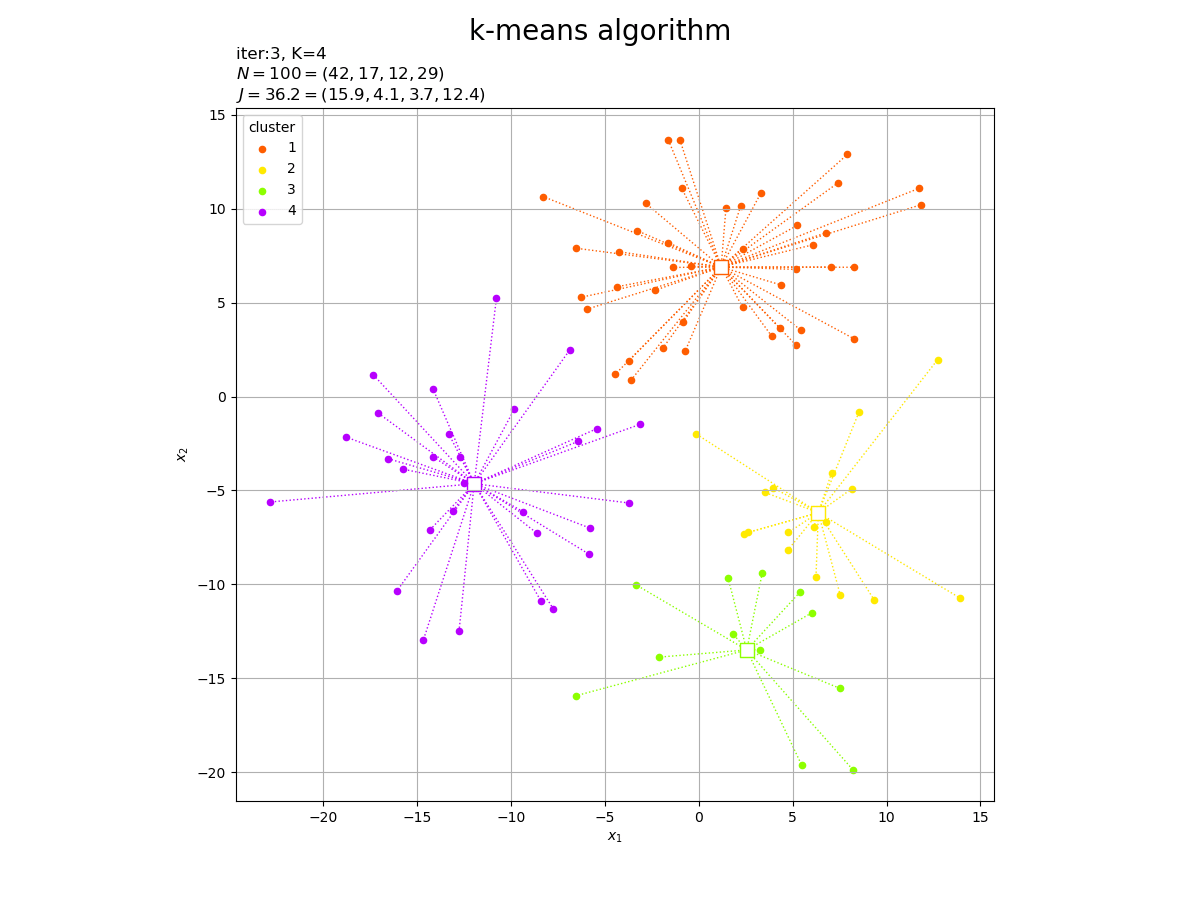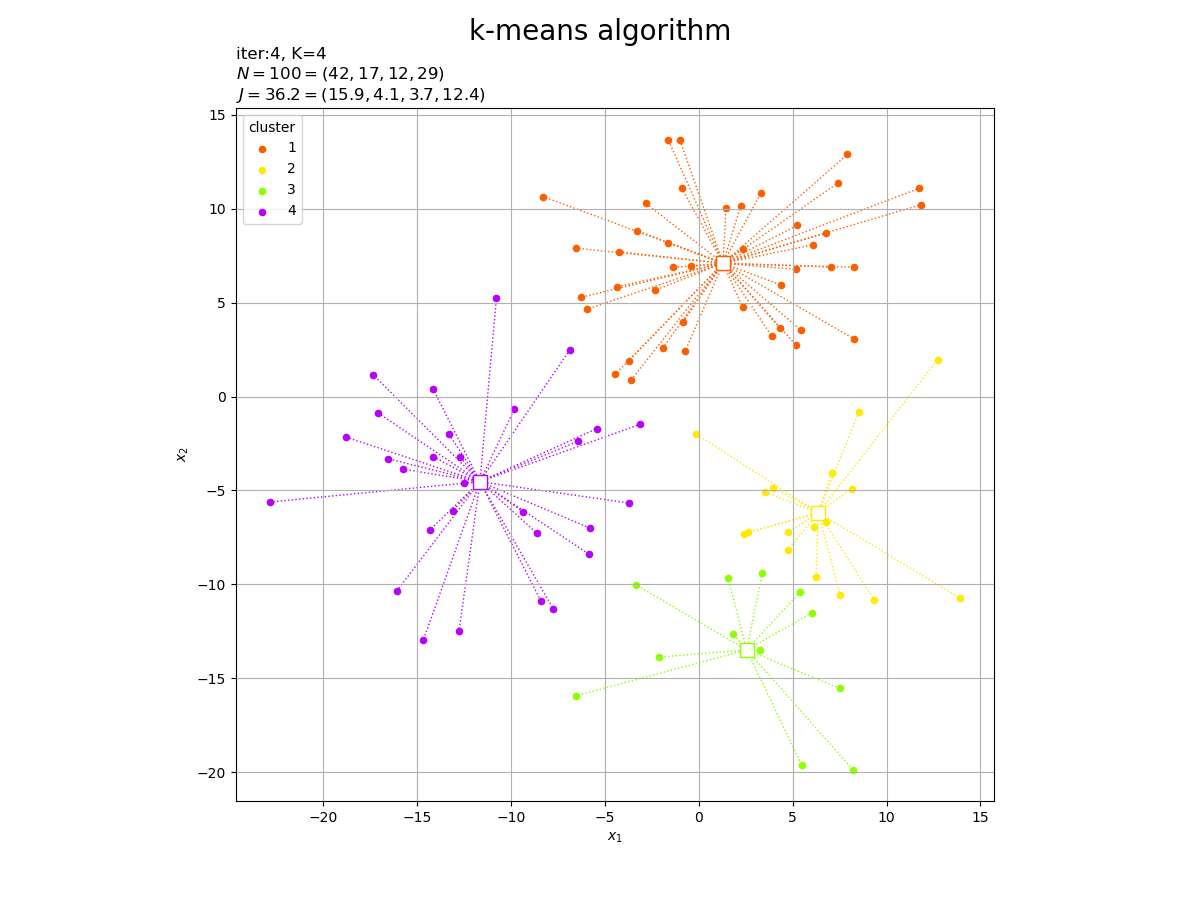
# カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(K) / K) # 2Dベクトルのクラスタを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white') for j in range(K): G_j, = np.where(c == j) # クラスタjのデータのインデック ax.plot([Z[j, 0].repeat(len(G_j)), X[G_j, 0]], [Z[j, 1].repeat(len(G_j)), X[G_j, 1]], color=colors[j], linewidth=1, linestyle=':', zorder=0) # 対応線 ax.scatter(x=Z[j, 0], y=Z[j, 1], edgecolor=colors[j], facecolor='white', marker='s', s=100) # 代表値 ax.scatter(x=X[G_j, 0], y=X[G_j, 1], color=colors[j], s=20, label=str(j+1)) # サンプルデータ ax.set_title('iter:' + str(cnt) + ', ' + 'N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + '), ' + 'J=' + str(np.sum(J).round(1)) + '=(' + ', '.join(map(str, J.round(1))) + ')', loc='left') fig.suptitle('k-means algorithm', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.grid() ax.legend(title='cluster') ax.set_aspect('equal') plt.show()
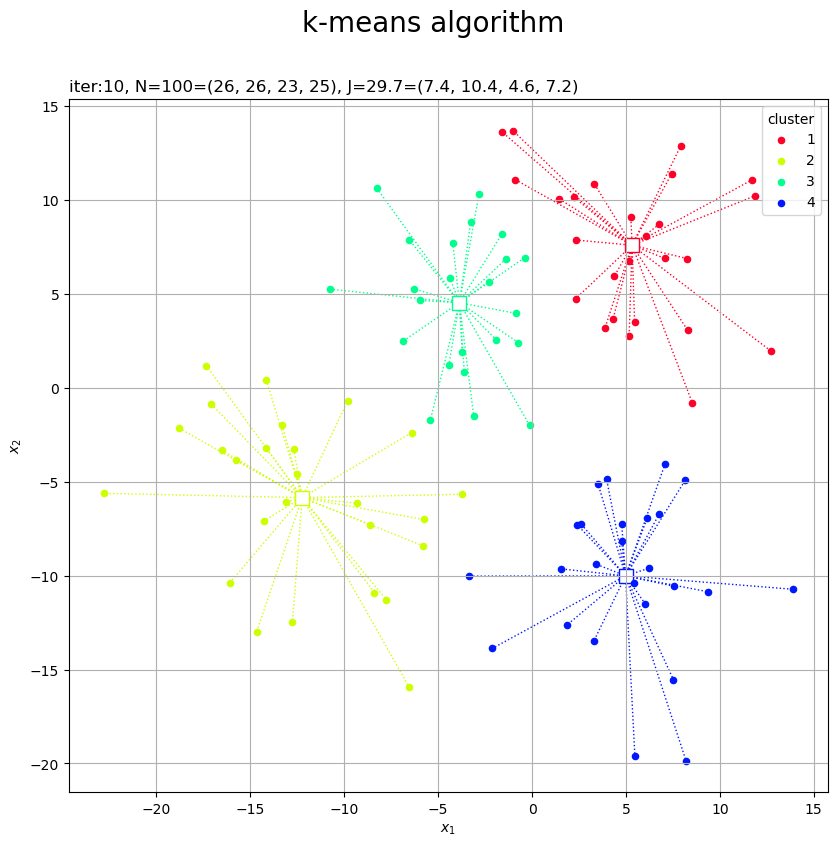
これまでと同様にして作図します。
目的関数の推移をグラフで確認します。
# 目的関数の推移を作図 fig, ax = plt.subplots(figsize=(8, 6), facecolor='white') ax.plot(np.arange(cnt+1), [np.sum(J) for J in trace_J]) # 目的関数 ax.set_xlabel('iteration') ax.set_ylabel('$J^{clust}$') ax.set_title('N=' + str(N) + ', K=' + str(K), loc='left') fig.suptitle('objective function', fontsize=20) ax.grid() plt.show()
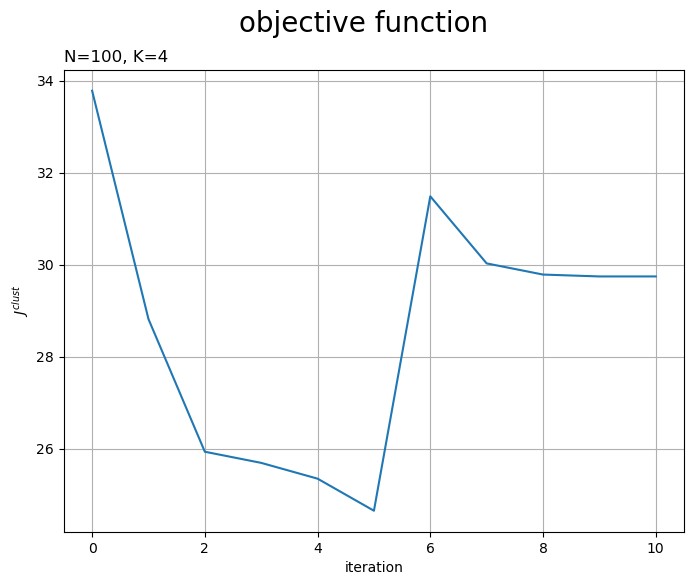
試行の度に目的関数が下がるのを確認できます。ただし、クラスタ数の変更時には値が上がることがあります。
更新の様子をアニメーションで確認します。
・作図コード(クリックで展開)
# フレーム数を設定 frame_num = cnt # カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(len(trace_Z[0])) / len(trace_Z[0])) # クラスタ数の削減に応じて色を入れ替え colors_lt = [colors] for i in range(frame_num): colors = colors[np.argsort(-np.int16(trace_G[i] >= G_num_lower))] # 継続するクラスタ番号の色を前に出す colors_lt.append(colors) # グラフオブジェクトを初期化 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white') fig.suptitle('k-means algorithm', fontsize=20) # 作図処理を関数として定義 def update(i): # 前フレームのグラフを初期化 plt.cla() # i回目の値を取得 Z = trace_Z[i] c = trace_c[i] G_num = trace_G[i] J = trace_J[i] colors = colors_lt[i] # クラスタ数を再設定 K = len(Z) # 2Dベクトルのクラスタを作図 for j in range(K): G_j, = np.where(c == j) # クラスタjのデータインデック ax.plot([Z[j, 0].repeat(len(G_j)), X[G_j, 0]], [Z[j, 1].repeat(len(G_j)), X[G_j, 1]], color=colors[j], linewidth=1, linestyle=':', zorder=0) # 対応線 ax.scatter(x=Z[j, 0], y=Z[j, 1], edgecolor=colors[j], facecolor='white', marker='s', s=100) # 代表値 ax.scatter(x=X[G_j, 0], y=X[G_j, 1], color=colors[j], s=20, label=str(j+1)) # サンプルデータ ax.set_title('iter:' + str(i) + ', ' + 'K=' + str(K) + '\n' + '$N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + ')$\n' + '$J=' + str(np.sum(J).round(1)) + '=(' + ', '.join(map(str, J.round(1))) + ')$', loc='left') ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.grid() ax.legend(title='cluster', loc='upper left') ax.set_aspect('equal') # gif画像を作成 ani = FuncAnimation(fig=fig, func=update, frames=frame_num, interval=300) # gif画像を保存 ani.save('k_means_gaussian_2d.gif')
作図処理をupdate()として定義して、FuncAnimation()でgif画像を作成します。
3次元の場合
次は、3次元空間上のベクトル(3次元のデータ)のクラスタリングを可視化します。
トイデータの生成
クラスタリングを行う3次元混合ガウス分布の乱数を生成します。
・作図コード(クリックで展開)
データの生成分布を設定します。
# 次元数を指定:(固定) D = 3 # 真のクラスタ数を指定 K_truth = 3 # K個の平均ベクトルを指定 mu = np.array( [[0.0, -3.0, -3.0], [3.0, 3.0, 0.0], [-3.0, 3.0, 3.0]] ) # K個の分散共分散行列を指定 sigma = np.array( [np.identity(D) * 3.0, np.identity(D) * 3.0, np.identity(D) * 3.0] ) # 混合比率を指定 pi = np.array([0.45, 0.25, 0.3])
次元数をとして、2次元のときと同様に、混合ガウス分布のパラメータと混合比率(クラスタの割り当て確率)を指定します。
クラスタの平均ベクトルと分散共分散行列はそれぞれ次の形状です。
各データに真のクラスタを割り当てます。
# データ数を指定 N = 100 # 真のクラスタを生成 c_truth_onehot = np.random.multinomial(n=1, pvals=pi, size=N) print(c_truth_onehot[:5]) # 真のクラスタ番号を抽出 _, c_truth = np.where(c_truth_onehot == 1) print(c_truth[:5]) # クラスタごとのデータ数を集計 G_num = np.sum(c_truth_onehot, axis=0) print(G_num)
[[1 0 0]
[0 1 0]
[0 0 1]
[0 0 1]
[0 1 0]]
[0 1 2 2 1]
[42 24 34]
「2次元の場合」と同じコードで、データごとに真のクラスタ、
を生成します。
データ(ベクトル)を生成します。
# データを生成 X = np.array([ np.random.multivariate_normal(mean=mu[j], cov=sigma[j], size=1).flatten() for j in c_truth ]) print(X[:5])
[[-0.9061569 -3.07634996 -2.36736738]
[ 3.83759876 3.80356171 2.01786429]
[-0.0631905 2.22261667 1.1232106 ]
[-0.01111482 4.86771311 0.96188149]
[ 7.40436435 1.09319964 0.55241717]]
「2次元の場合」と同じコードで、割り当てられたクラスタに応じてデータ、
を生成します。
データと真のクラスタを散布図で確認します。
# 目的関数(ノルムの2乗平均)を計算 J_truth = np.array( [np.sum(np.linalg.norm(X[c_truth == j] - mu[j], axis=1)**2) / N for j in range(K_truth)] ) # カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(K_truth) / K_truth) # サイズ用の値を設定 z_min = X[:, 2].min() + 1 z_max = X[:, 2].max() + 1 # 3Dベクトルの真のクラスタを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white', subplot_kw={'projection': '3d'}) for j in range(K_truth): G_j, = np.where(c_truth == j) # クラスタjのデータインデック ax.quiver(*np.tile(mu[j], reps=(len(G_j), 1)).T, *(X[G_j]-mu[j]).T, color=colors[j], arrow_length_ratio=0.0, linewidth=0.5, linestyle=':') # 対応線 ax.scatter(*mu[j].T, edgecolor=colors[j], facecolor='white', marker='s', s=100, zorder=1) # 平均値 ax.scatter(*X[G_j].T, color=colors[j], s=20, label=str(j+1), zorder=2) # サンプルデータ ax.quiver(mu[:, 0], mu[:, 1], mu[:, 2], np.zeros(K_truth), np.zeros(K_truth), z_min-mu[:, 2], color='gray', arrow_length_ratio=0, linestyle=':') # 補助線 ax.set_title('N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + '), ' + 'J=' + str(np.sum(J_truth).round(1)) + '=(' + ', '.join(map(str, J_truth.round(1))) + ')', loc='left') fig.suptitle('true cluster', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.set_zlabel('$x_3$') ax.legend(title='cluster') ax.set_zlim(z_min, z_max) plt.show()
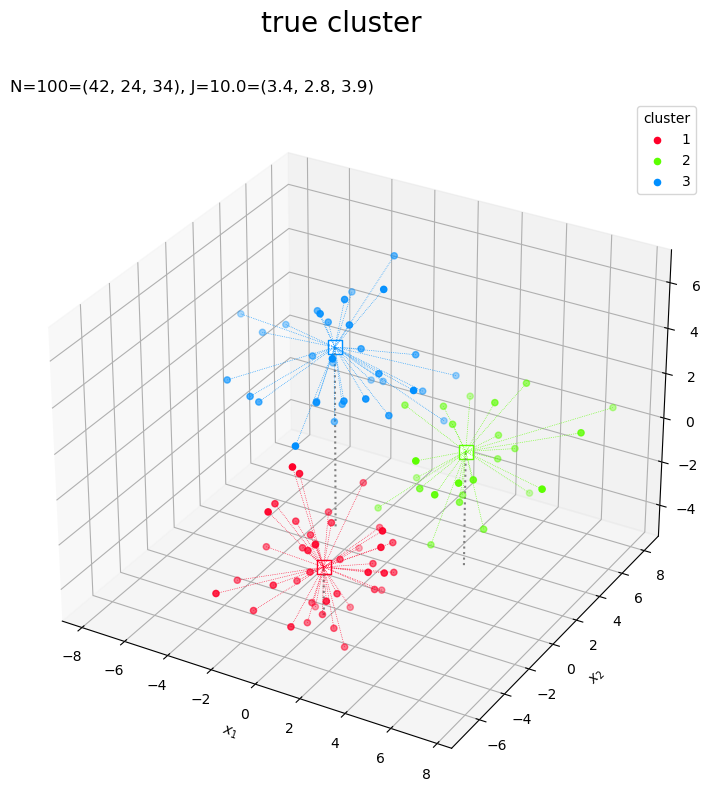
各データを塗りつぶしの丸、真の平均値を白抜きの四角として描画します。
クラスタごとの代表値と各データを結ぶ線分を(3Dプロットだとaxes.plot()でまとめて描画できなかった?ので)axes.quiver()で描画します。第1・2・3引数に始点の座標、第4・5・6引数に変化量(ベクトルのサイズ)を指定します。
それぞれ、配列の前に*を付けてアンパック(展開)して指定しています。
以上で、クラスタリングに利用する人工データを作成できました。
クラスタリング
3次元ベクトルに対するk平均法を実装します。
2通りの方法で初期値を設定できます。
1つ目は、クラスタの初期値をランダムに割り当てて、クラスタごとに代表値として平均値を計算します。
# クラスタ数の初期値を指定 K = 10 # ランダムにクラスタを割り当て c_onehot = np.random.multinomial(n=1, pvals=np.repeat(1, K)/K, size=N) print(c_truth_onehot[:5]) # クラスタ番号を抽出 _, c = np.where(c_onehot == 1) print(c[:5]) # クラスタごとのデータ数を集計 G_num = np.sum(c_onehot, axis=0) print(G_num) # クラスタの代表値(平均値)を計算 Z = np.array( [np.mean(X[c == j], axis=0) for j in range(K)] ) print(Z[:5]) # 目的関数(ノルムの2乗平均)を計算 J = np.array( [np.sum(np.linalg.norm(X[c == j] - Z[j], axis=1)**2) / N for j in range(K)] ) print(J[:5])
[[0 0 0 0 1 0 0 0 0 0]
[0 0 0 0 1 0 0 0 0 0]
[0 0 0 0 0 0 0 0 1 0]
[0 0 0 0 0 0 0 1 0 0]
[0 0 1 0 0 0 0 0 0 0]]
[5 5 8 7 2]
[ 8 2 10 10 9 17 10 16 7 11]
[[-0.83196199 -0.71145777 -0.20413865]
[-1.80777768 -2.11801431 -0.68183497]
[ 0.98736262 1.46167339 0.72020464]
[ 0.06203712 -0.47859545 -1.06351002]
[-1.0299771 0.71466154 0.25949943]]
[1.55461591 0.29448437 2.69100556 2.68898349 1.96192039]
「2次元の場合」と同じコードで、クラスタ、
と代表値
、
を初期化します。
真のクラスタのときと同様にして、クラスタの初期値をグラフで確認します。
# カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(K) / K) # サイズ用の値を設定 z_min = X[:, 2].min() + 1 z_max = X[:, 2].max() + 1 # 3Dベクトルのクラスタを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white', subplot_kw={'projection': '3d'}) for j in range(K): G_j, = np.where(c == j) # クラスタjのデータインデック ax.quiver(*np.tile(Z[j], reps=(len(G_j), 1)).T, *(X[G_j]-Z[j]).T, color=colors[j], arrow_length_ratio=0.0, linewidth=0.5, linestyle=':') # 対応線 ax.scatter(*Z[j].T, edgecolor=colors[j], facecolor='white', marker='s', s=100, zorder=1) # 平均値 ax.scatter(*X[G_j].T, color=colors[j], s=20, label=str(j+1), zorder=2) # サンプルデータ ax.quiver(Z[:, 0], Z[:, 1], Z[:, 2], np.zeros(K), np.zeros(K), z_min-Z[:, 2], color='gray', arrow_length_ratio=0, linestyle=':') # 補助線 ax.set_title('iter:0, ' + 'N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + '), ' + 'J=' + str(np.sum(J).round(1)) + '=(' + ', '.join(map(str, J.round(1))) + ')', loc='left') fig.suptitle('initial cluster', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.set_zlabel('$x_3$') ax.legend(title='cluster') ax.set_zlim(z_min, z_max) ax.set_aspect('equal') plt.show()
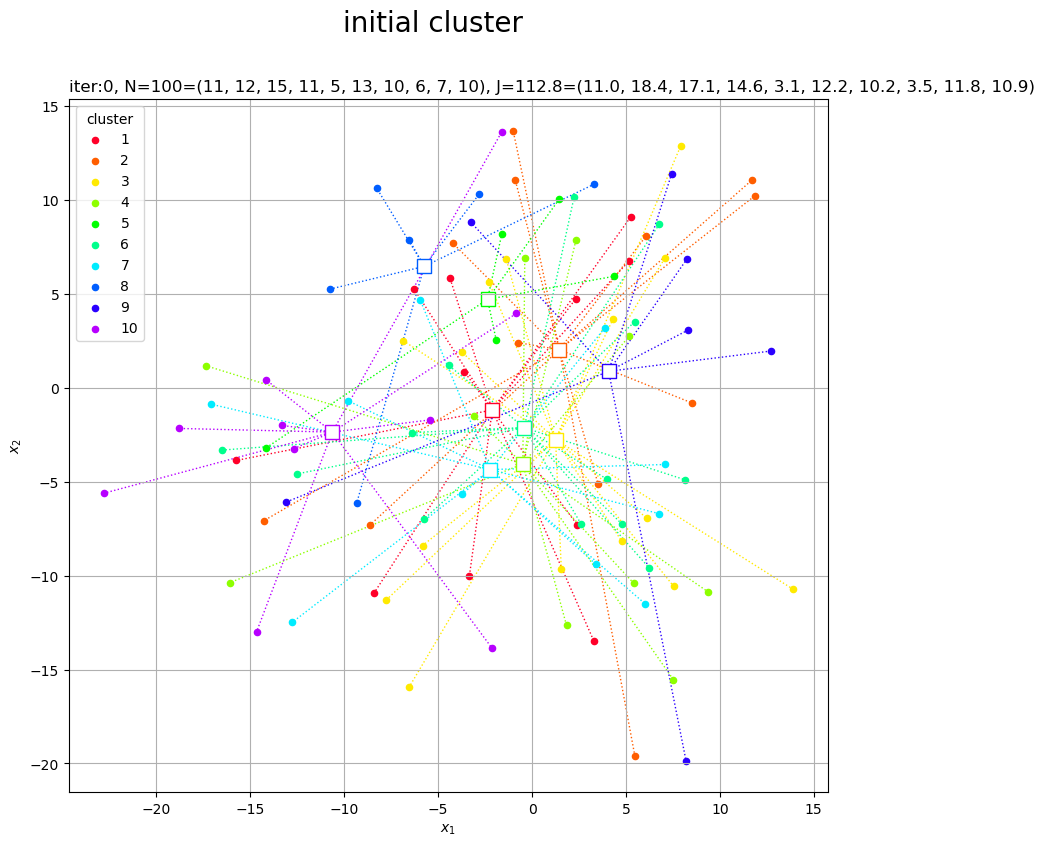
ランダムに割り当てられたデータの平均値なので、代表値がデータ全体の中心付近に集まっています。
2つ目は、代表値をランダムに設定して、データごとに代表値との距離が近い(ノルムが小さい)クラスタを割り当てます。
# クラスタ数の初期値を指定 K = 10 # ランダムにクラスタの代表値を設定 Z = np.array( [np.random.uniform(low=X[:, 0].min(), high=X[:, 0].max(), size=K), np.random.uniform(low=X[:, 1].min(), high=X[:, 1].max(), size=K), np.random.uniform(low=X[:, 2].min(), high=X[:, 2].max(), size=K)] ).T print(Z[:5]) # ノルムが最小のクラスタを割り当て c = np.argmin( [np.linalg.norm(X - Z[j], axis=1) for j in range(K)], axis=0 ) print(c[:5]) # クラスタごとのデータ数を集計 G_num = np.array( [np.sum(c == j) for j in range(K)] ) print(G_num) # 目的関数(ノルムの2乗平均)を計算 J = np.array( [np.sum(np.linalg.norm(X[c == j] - Z[j], axis=1)**2) / N for j in range(K)] ) print(J[:5])
[[ 3.22789816 5.73999796 3.33690734]
[-1.28032404 -0.79235536 -4.94935583]
[-1.14602146 1.86271125 -3.97244374]
[ 1.28276238 1.73445996 -4.20285614]
[ 6.29835174 -3.55930852 1.68244032]]
[1 0 2 0 9]
[29 16 18 10 0 10 15 1 0 1]
[7.89345639 2.37520645 7.21887148 0.94524146 0. ]
代表値Zとして、K行3列の一様乱数を生成します。
それ以外は「2次元の場合」と同じコードで初期化できます。
1つ目の方法と同じコードで、クラスタの初期値をグラフで確認します。
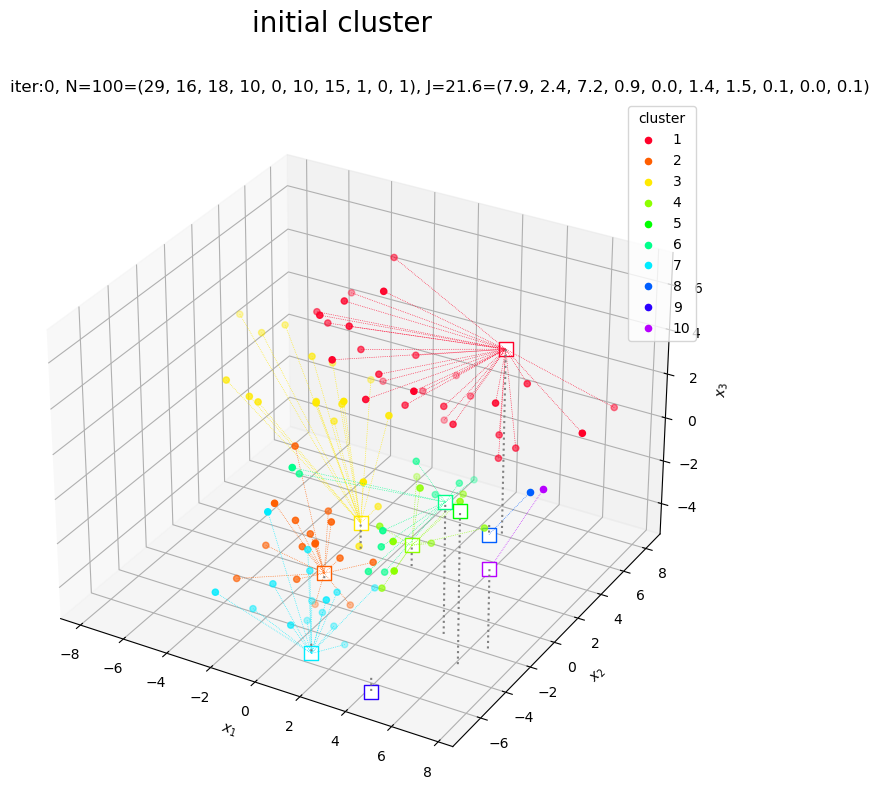
代表値がばらけるので、ある程度まとまったクラスタができています。データが割り当てられないクラスタができることがあります。
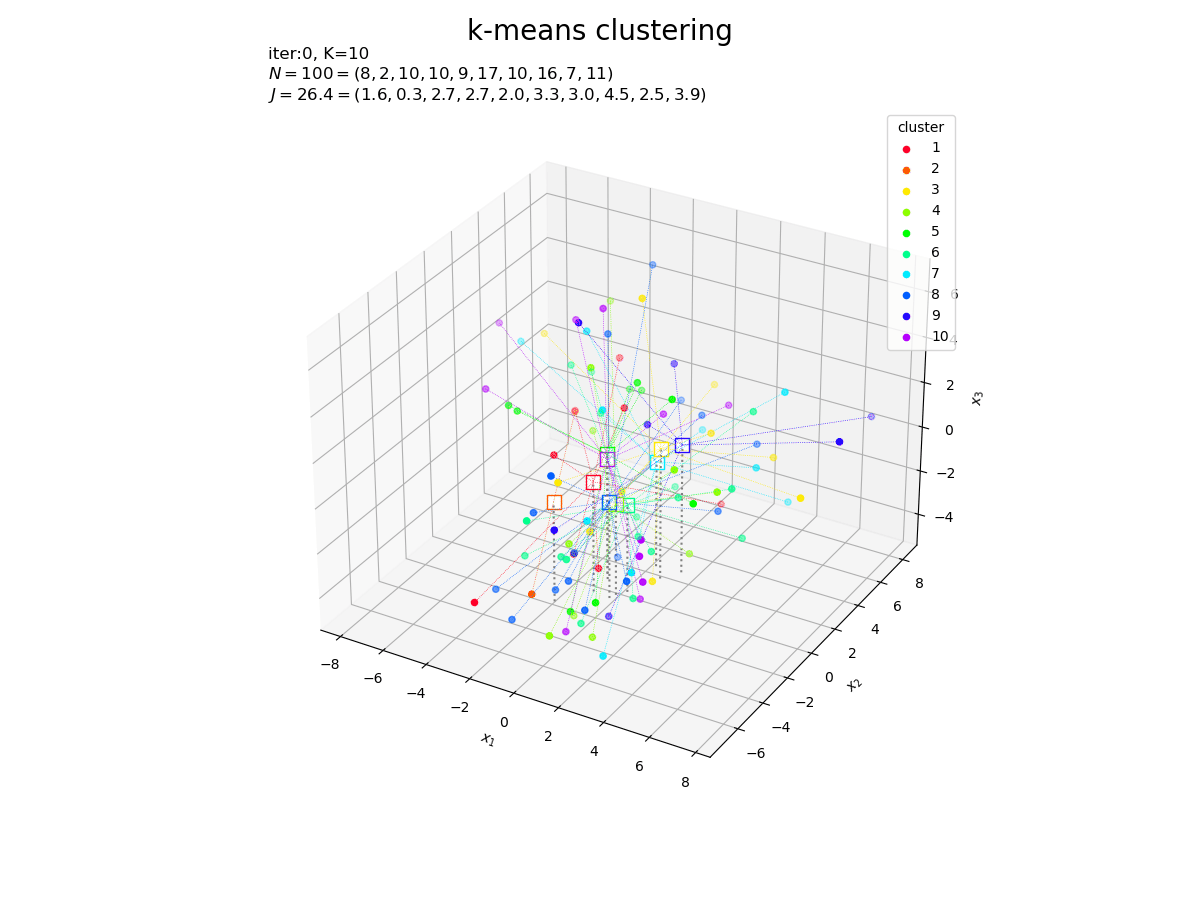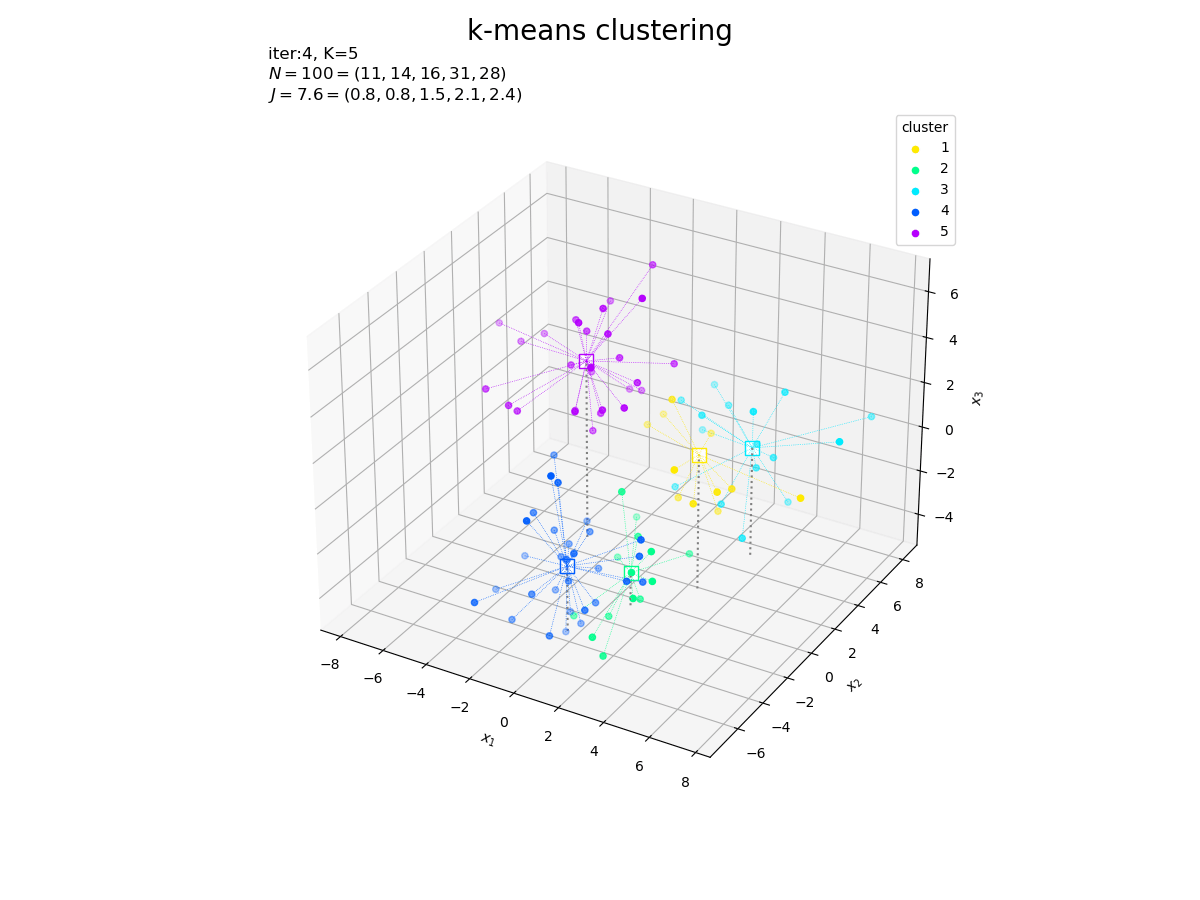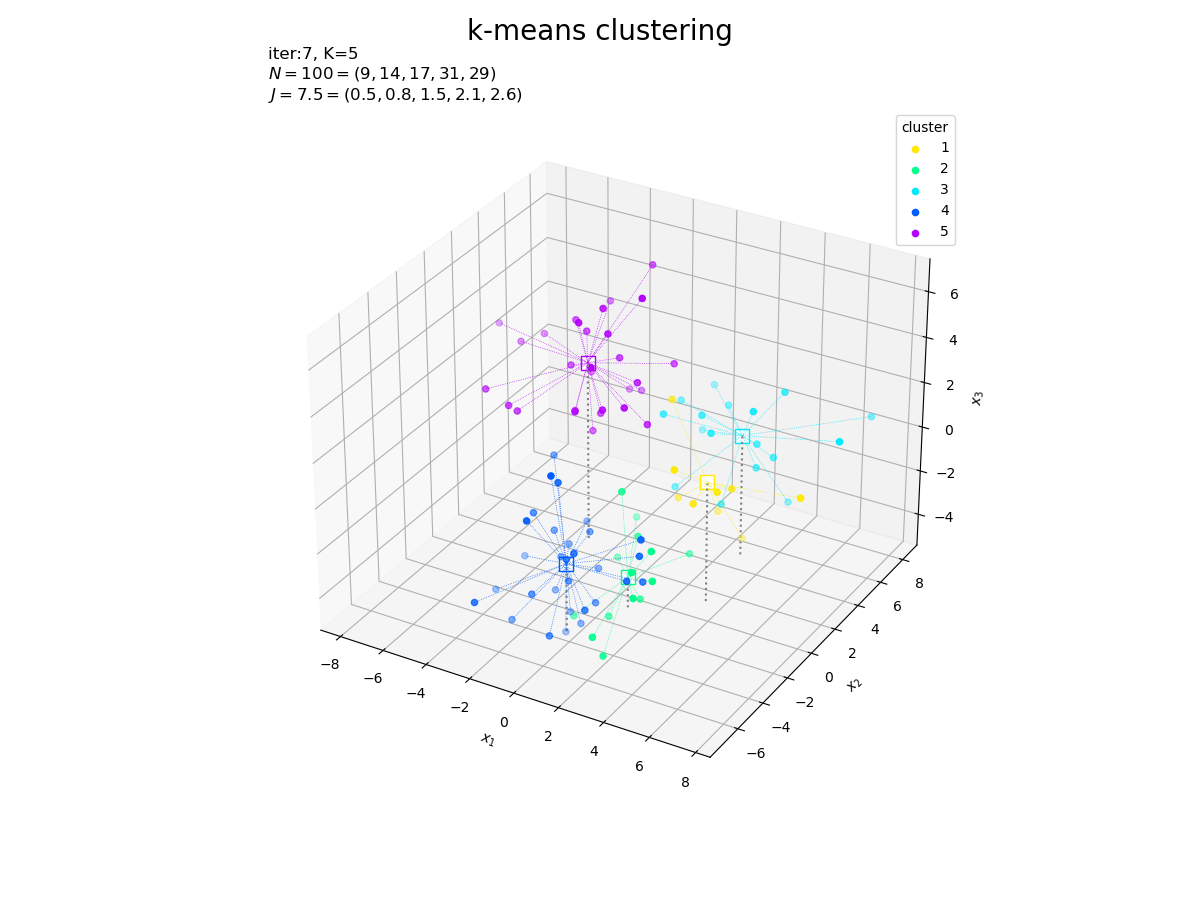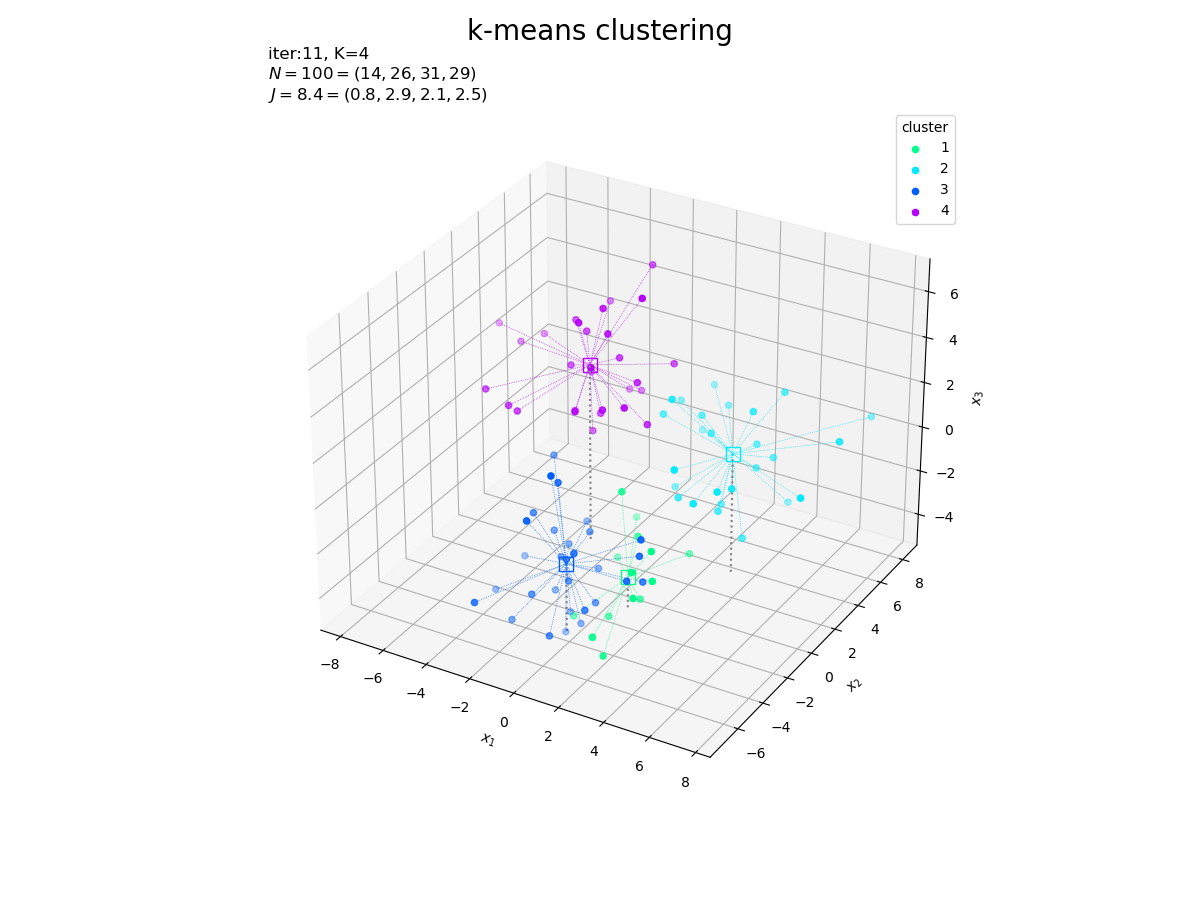
k平均法によりクラスタと代表値を繰り返し更新します。
--- iter:1 ---
K=6
|G|=[24 13 20 16 10 17]
J=8.92923
--- iter:2 ---
K=6
|G|=[21 17 22 16 8 16]
J=7.70315
--- iter:3 ---
K=5
|G|=[17 19 27 19 18]
J=8.02358
--- iter:4 ---
K=5
|G|=[18 19 27 17 19]
J=7.39068
--- iter:5 ---
K=5
|G|=[18 19 27 17 19]
J=7.35017
--- iter:6 ---
K=5
|G|=[18 19 27 17 19]
J=7.35017
「2次元の場合」のコードで処理できます。
クラスタの更新値(収束結果)をグラフで確認します。
# カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(K) / K) # サイズ用の値を設定 z_min = X[:, 2].min() + 1 z_max = X[:, 2].max() + 1 # 3Dベクトルのクラスタを作図 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white', subplot_kw={'projection': '3d'}) for j in range(K): G_j, = np.where(c == j) # クラスタkのデータインデック ax.quiver(*np.tile(Z[j], reps=(len(G_j), 1)).T, *(X[G_j]-Z[j]).T, color=colors[j], arrow_length_ratio=0.0, linewidth=0.5, linestyle=':') # 対応線 ax.scatter(*Z[j].T, edgecolor=colors[j], facecolor='white', marker='s', s=100, zorder=1) # 代表値 ax.scatter(*X[G_j].T, color=colors[j], s=20, label=str(j+1), zorder=2) # サンプルデータ ax.quiver(Z[:, 0], Z[:, 1], Z[:, 2], np.zeros(K), np.zeros(K), z_min-Z[:, 2], color='gray', arrow_length_ratio=0, linestyle=':') # 補助線 ax.set_title('iter:' + str(cnt) + ', ' + 'N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + '), ' + 'J=' + str(np.sum(J_truth).round(1)) + '=(' + ', '.join(map(str, J_truth.round(1))) + ')', loc='left') fig.suptitle('k-means clustering', fontsize=20) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.set_zlabel('$x_3$') ax.legend(title='cluster') ax.set_zlim(z_min, z_max) ax.set_aspect('equal') plt.show()
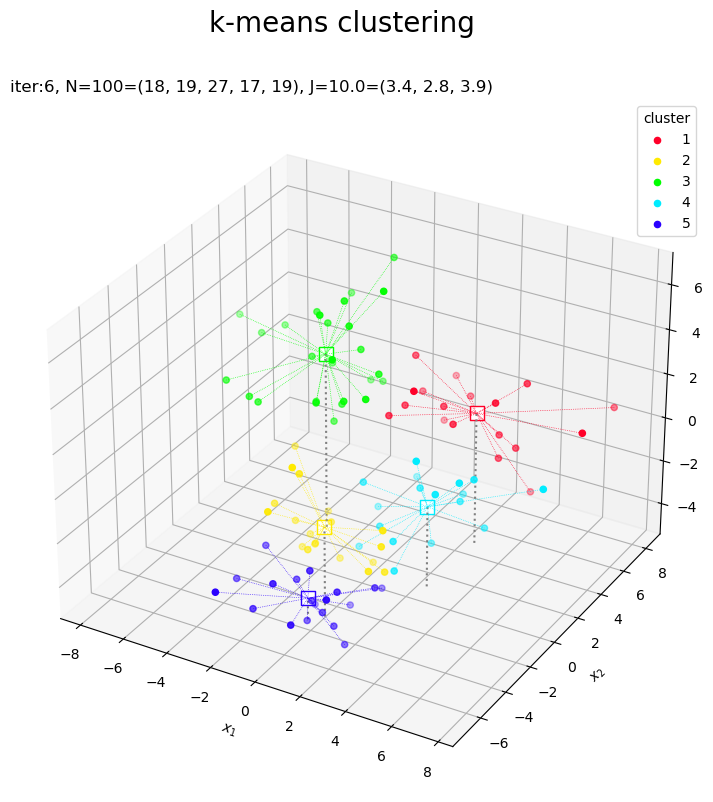
これまでと同様にして作図します。
目的関数の推移をグラフで確認します。
# 目的関数の推移を作図 fig, ax = plt.subplots(figsize=(8, 6), facecolor='white') ax.plot(np.arange(cnt+1), [np.sum(J) for J in trace_J]) # 目的関数 ax.grid() ax.set_xlabel('iteration') ax.set_ylabel('$J^{clust}$') ax.set_title('N=' + str(N) + ', K=' + str(K), loc='left') fig.suptitle('objective function', fontsize=20) plt.show()
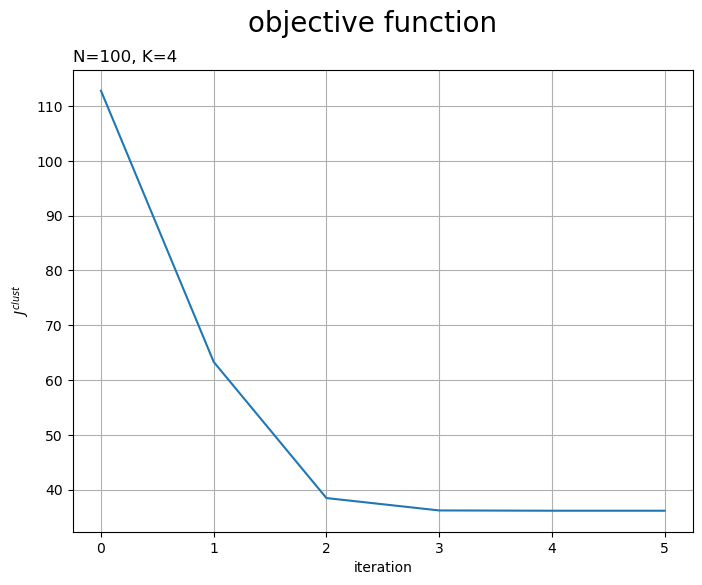
試行の度に目的関数が下がるのを確認できます。ただし、クラスタ数の変更時には値が上がることがあります。
更新の様子をアニメーションで確認します。
・作図コード(クリックで展開)
# フレーム数を設定 frame_num = cnt # カラーマップを指定 cm = plt.get_cmap('gist_rainbow') colors = cm(np.arange(len(trace_Z[0])) / len(trace_Z[0])) # クラスタ数の削減に応じて色を入れ替え colors_lt = [colors] for i in range(frame_num): colors = colors[np.argsort(-np.int16(trace_G[i] >= G_num_lower))] # 継続するクラスタ番号の色を前に出す colors_lt.append(colors) # サイズ用の値を設定 z_min = X[:, 2].min() + 1 z_max = X[:, 2].max() + 1 # グラフオブジェクトを初期化 fig, ax = plt.subplots(figsize=(12, 9), facecolor='white', subplot_kw={'projection': '3d'}) fig.suptitle('k-means clustering', fontsize=20) # 作図処理を関数として定義 def update(i): # 前フレームのグラフを初期化 plt.cla() # i回目の値を取得 Z = trace_Z[i] c = trace_c[i] G_num = trace_G[i] J = trace_J[i] colors = colors_lt[i] # クラスタ数を再設定 K = len(Z) # 3Dベクトルのクラスタを作図 for j in range(K): G_j, = np.where(c == j) # クラスタjのデータインデック ax.quiver(*np.tile(Z[j], reps=(len(G_j), 1)).T, *(X[G_j]-Z[j]).T, color=colors[j], arrow_length_ratio=0.0, linewidth=0.5, linestyle=':') # 対応線 ax.scatter(*Z[j].T, edgecolor=colors[j], facecolor='white', marker='s', s=100, zorder=1) # 代表値 ax.scatter(*X[G_j].T, color=colors[j], s=20, label=str(j+1), zorder=2) # サンプルデータ ax.quiver(Z[:, 0], Z[:, 1], Z[:, 2], np.zeros(K), np.zeros(K), z_min-Z[:, 2], color='gray', arrow_length_ratio=0, linestyle=':') # 補助線 ax.set_title('iter:' + str(i) + ', ' + 'K=' + str(K) + '\n' + '$N=' + str(N) + '=(' + ', '.join(map(str, G_num)) + ')$\n' + '$J=' + str(np.sum(J).round(1)) + '=(' + ', '.join(map(str, J.round(1))) + ')$', loc='left') ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.set_zlabel('$x_3$') ax.legend(title='cluster') ax.set_zlim(z_min, z_max) # gif画像を作成 ani = FuncAnimation(fig=fig, func=update, frames=frame_num, interval=300) # gif画像を保存 ani.save('k_means_gaussian_3d.gif')

この記事では、多次元混合ガウス分布の乱数に対するk平均法を実装しました。次の記事では、MNISTデータセットに対するk平均法を実装します。
参考書籍
- Stephen Boyd・Lieven Vandenberghe(著),玉木 徹(訳)『スタンフォード ベクトル・行列からはじめる最適化数学』講談社サイエンティク,2021年.
おわりに
アルゴリズム的な意味でほんとシンプルに実装できるんですね。
ただクラスタ数の更新周りをももう少しなんとかしてみたいです。今回は雑にクラスタを削減しました。試行回数に応じて削除基準を変更してみたいです。他に、近い代表値を統合するのもよさそうな気がします。(が、別にしなくてもいいですよね。)
これまでにシンプルじゃない方法もやってきました。こっちも面白いので覗いてみてください。
【次の内容】