はじめに
『パターン認識と機械学習』の独学時のまとめです。一連の記事は「数式の行間埋め」または「R・Pythonでのスクラッチ実装」からアルゴリズムの理解を補助することを目的としています。本とあわせて読んでください。
この記事は、4.3.4項の内容です。多クラスロジスティック回帰をPythonで実装します。
【数式読解編】
【前節の内容】
【他の節一覧】
【この節の内容】
4.3.4 多クラスロジスティック回帰の実装:入力が2次元の場合
最急降下法によるロジスティック回帰の最尤推定を実装します。
利用するライブラリを読み込みます。
# 4.3.4項で利用するライブラリ import numpy as np from scipy.stats import multivariate_normal # 多次元ガウス分布 import matplotlib.pyplot as plt from matplotlib.animation import FuncAnimation
推定過程をアニメーション(gif画像)で可視化するのにMatplotlibライブラリのanimationモジュールを利用します。不要であれば省略してください。
・利用する関数の作成
推定に利用する関数を作成します。基底関数については「【Python】3.1.0:基底関数の作図【PRMLのノート】 - からっぽのしょこ」を、計画行列については「【Python】3.1.1:線形基底関数モデルの実装【PRMLのノート】 - からっぽのしょこ」を参照してください。
ロジスティックシグモイド関数を実装します。
# ロジスティックシグモイド関数を作成 def sigmoid(x): return 1.0 / (1.0 + np.exp(-x))
ロジスティックシグモイド関数は、次の式で定義されます。
ソフトマックス関数を実装します。
# ソフトマックス関数の実装 def softmax(x_k): # 最大値を引く:オーバーフロー対策 x_k -= np.max(x_k, axis=-1, keepdims=True) # ソフトマックス関数の計算 return np.exp(x_k) / np.sum(np.exp(x_k), axis=-1, keepdims=True)
ソフトマックス関数は、次の式で定義されます。
ただし、オーバーフローを回避する処理を行っています。
非線形変換を行う基底関数$\phi_j(\mathbf{x})$を用いた計画行列
の計算を行う関数を作成します。この例では、0番目の基底関数について$\phi_0(\mathbf{x}_1) = 1$とします。
多項式基底関数の計画行列を定義します。
ガウス基底関数の計画行列を定義します。
シグモイド基底関数の計画行列を定義します。
# 2次元シグモイド基底関数を作成 def phi_sigmoid2d(x_d, mu_d, s_d): # m番目のパラメータにより入力値を調整 a_d = (x_d - mu_d) / s_d # ロジスティックシグモイド関数の計算 y_d = sigmoid(a_d) # データごとに総和を計算 return np.sum(y_d, axis=1) / len(s_d) # 2次元シグモイド基底関数の計画行列を作成:(対角線に標準化) def Phi_sigmoid2d(x_nd, M, _x_nd=None): # パラメータ設定用の入力値を設定 if _x_nd is None: _x_nd = x_nd.copy() # M-1個のパラメータを作成 s_d = np.std(_x_nd, axis=0) # 標準偏差で固定 if M == 2: # 入力値の平均 mu_md = np.array([[np.mean(_x_nd[:, 0]), np.mean(_x_nd[:, 1])]]) elif M == 3: # 調整幅を指定 sgm_d = s_d * 2.0 # 入力値の平均 mu_md = np.array( [[np.mean(_x_nd[:, 0]) - sgm_d[0], np.mean(_x_nd[:, 0]) - sgm_d[1]], [np.mean(_x_nd[:, 0]) + sgm_d[0], np.mean(_x_nd[:, 0]) + sgm_d[1]]] ) elif M > 3: # 調整幅を指定 sgm_d = s_d * 0.5 # 最小値から最大値を等分 mu_md = np.stack([ np.linspace(np.min(_x_nd[:, 0]) + sgm_d[0], np.max(_x_nd[:, 0] - sgm_d[1]), num=M-1), np.linspace(np.min(_x_nd[:, 1]) + sgm_d[0], np.max(_x_nd[:, 1] - sgm_d[1]), num=M-1) ], axis=1) # 変数を初期化 phi_x_nm = np.ones((len(x_nd), M)) # 2次元シグモイド基底関数による変換 for m in range(1, M): phi_x_nm[:, m] = phi_sigmoid2d(x_nd, mu_md[m-1], s_d) return phi_x_nm
多項式基底関数とガウス基底関数については「【Python】4.3.2-3:ロジスティック回帰の実装:入力が2次元の場合【PRMLのノート】 - からっぽのしょこ」を参照してください。
(2次元の基底関数を調べても見付けられなかったので、雰囲気で実装しました。正しい情報を教えて下さい。)
・データの生成
推定に利用するデータを人工的に作成します。
データ生成に関するパラメータを指定します。ここでは、入力が2次元の場合なので$D = 2$です。
# クラス数を指定 K = 3 # クラス割り当て確率を指定 pi_k = np.array([0.4, 0.35, 0.25]) # データ生成用のK個の平均を指定 mu_kd = np.array( [[-2.5, 1.5], [0.0, 2.0], [2.5, 2.5]] ) # データ生成用のK個の分散共分散行列を指定 sigma2_kdd = np.array( [[[0.25, 0.2], [0.2, 0.25]], [[0.25, -0.2], [-0.2, 0.25]], [[0.25, 0.2], [0.2, 0.25]]] )
クラス$k$($t_{n,k} = 1$)となる確率を$\pi_k$として、各クラスの割り当て確率を$K$次元ベクトル$\boldsymbol{\pi} = (\pi_1, \cdots, \pi_K)$で表します。$0 \leq \pi_k \leq 1$、$\sum_{k=1}^K \pi_k = 1$となるように1次元配列pi_kに値を指定します。
クラス$k$の平均ベクトルを$D$次元ベクトル$\boldsymbol{\mu}_k = (\mu_{k,1}, \mu_{k,2})$で表します。$K$個の平均ベクトルをまとめた$\boldsymbol{\mu} = \{\boldsymbol{\mu}_1, \cdots, \boldsymbol{\mu}_K\}$を、2次元配列mu_kdとします。
同様に、クラス$k$の分散共分散行列を$D \times D$の行列$\boldsymbol{\Sigma}_k = (\sigma_{k,1,1}^2, \cdots, \sigma_{k,2,2}^2)$で表します。$K$個の分散共分散行列をまとめた$\boldsymbol{\Sigma} = \{\boldsymbol{\Sigma}_1, \cdots, \boldsymbol{\Sigma}_K\}$を、3次元配列sigma2_kddとします。
指定したパラメータに従って、真のクラスを作成します。
# データ数を指定 N = 250 # 真のクラスを生成 t_nk = np.random.multinomial(n=1, pvals=pi_k, size=N) print(t_nk[:5]) # 真のクラス番号を抽出 _, t_n = np.where(t_nk == 1) print(t_n[:5])
[[1 0 0]
[0 1 0]
[1 0 0]
[1 0 0]
[0 1 0]]
[0 1 0 0 1]
データ数$N$を指定して、観測データ(目標値$\mathbf{T} = \{\mathbf{t}_1, \cdots, \mathbf{t}_N\}$、入力値$\mathbf{X} = \{\mathbf{x}_1, \cdots, \mathbf{x}_N\}$)を作成します。
$\mathbf{t}_n = (t_{n,1}, \cdots, t_{n,K})$は$n$番目のデータのクラスを表し、$t_{n,k} = 1$であればクラス$k$とします。
対応する入力($n$番目の入力)は$D$次元ベクトル$\mathbf{x}_n = (x_{n,1}, x_{n,2})$です。
$\mathbf{t}$はベルヌーイ分布に従い生成します。ベルヌーイ分布に従う乱数は、二項分布の乱数生成関数np.random.binomial()の試行回数の引数nに1を指定することで生成できます。確率の引数pにクラスの割り当て確率pi_k、データ数(サンプルサイズ)の引数sizeにNを指定します。
生成した真のクラスに従って、入力値を作成します。
# 真のクラスに従いデータを生成 x_nd = np.array( [np.random.multivariate_normal(mean=mu_kd[k], cov=sigma2_kdd[k], size=1).flatten() for k in t_n] ) print(x_nd[:5])
[[-3.2336972 0.89190788]
[ 0.44406785 1.46138796]
[-2.54989025 1.63059581]
[-2.47420479 1.63554913]
[-0.59805095 2.76553544]]
$\mathbf{X}$は2次元ガウス分布に従い生成します。多次元ガウス分布に従う乱数は、np.random.multivariate_normal()で生成できます。meanは平均、covは分散共分散行列の引数です。データごとに割り当てられたクラスに応じてパラメータを指定する必要があります。
クラスに対応したパラメータの値は、次のようにして取り出しています。
# パラメータの取得 print(t_n[:5]) print(mu_kd[t_n[:5]])
[0 1 0 0 1]
[[-2.5 1.5]
[ 0. 2. ]
[-2.5 1.5]
[-2.5 1.5]
[ 0. 2. ]]
t_nには各データのクラスが保存されています。
クラス$k$のパラメータはmu_kd[k], sigma2_kdd[k]なので、t_nの値がインデックスに対応します。
作図用と計算用の$\mathbf{x}$の値を作成します。
# 作図用のxの値を作成 x1 = np.linspace(np.min(x_nd[:, 0]) - 0.5, np.max(x_nd[:, 0]) + 0.5, num=250) x2 = np.linspace(np.min(x_nd[:, 1]) - 0.5, np.max(x_nd[:, 1]) + 0.5, num=250) # 作図用のxの点を作成 X1, X2 = np.meshgrid(x1, x2) # 計算用のxの点を作成 x_points = np.stack([X1.flatten(), X2.flatten()], axis=1) x_dims = X1.shape print(x_points.shape) print(x_dims)
(62500, 2)
(250, 250)
次元ごとに最小値から最大値までの値x1, x2を作成します。
x1, x2を格子状の点に変換して、作図用の値X1, X2とします。
X1, X2をそれぞれ1列に並べて、計算用の値x_pointsとします。
データの確認用に、入力$\mathbf{x}$の分布を計算します。
# 混合ガウス分布を計算 model_density = 0.0 for k in range(K): # クラスkの確率密度を加算 model_density += pi_k[k] * multivariate_normal.pdf(x=x_points, mean=mu_kd[k], cov=sigma2_kdd[k]) print(np.round(model_density[:5], 5))
[0.0002 0.00023 0.00024 0.00026 0.00028]
混合ガウス分布の確率密度は、次の式で計算します。
クラスごとの分布$\mathcal{N}(\mathbf{x} | \boldsymbol{\mu}_k, \boldsymbol{\sigma}_k)$を、各クラスとなる確率$\pi_k$で加重平均を求めています。
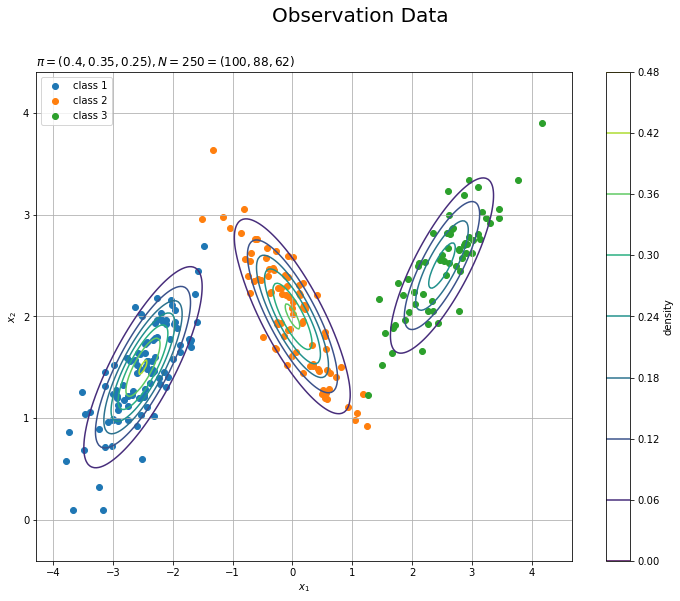
観測データの散布図を混合分布に重ねて描画します。
# 観測モデルを作図 plt.figure(figsize=(12, 9)) for k in range(K): k_idx = t_n == k plt.scatter(x=x_nd[k_idx, 0], y=x_nd[k_idx, 1], label='class ' + str(k+1)) # 各クラスの観測データ plt.contour(X1, X2, model_density.reshape(x_dims)) # データ生成分布 plt.xlabel('$x_1$') plt.ylabel('$x_2$') plt.suptitle('Observation Data', fontsize=20) plt.title('$\pi=(' + ', '.join([str(pi) for pi in pi_k]) + ')' + ', N=' + str(N) + '=(' + ', '.join([str(np.sum(t_n == k)) for k in range(K)]) + ')$', loc='left') plt.colorbar(label='density') plt.legend() plt.grid() plt.show()
・最急降下法による最尤推定
最急降下法によりロジスティック回帰のパラメータの最尤解を求めます。
・処理の解説
まずは、繰り返し行う個々の処理を解説します。次に、それらをまとめて推定を行います。
基底関数を設定して、計画行列の計算を行い入力値を変換します。
# 基底関数の数を指定 M = 3 # 基底関数を指定 #phi = Phi_polynomial2d #phi = Phi_gauss2d phi = Phi_sigmoid2d # 基底関数により入力値を変換 phi_x_nm = phi(x_nd, M) print(phi_x_nm[:5])
[[1. 0.81592234 0.25977638]
[1. 0.95450739 0.43043086]
[1. 0.85780228 0.39484159]
[1. 0.86147454 0.3963635 ]
[1. 0.93431699 0.5256328 ]]
利用する基底関数の計画行列Phi_***()を関数phi()として複製します。
phi()を使って計算します。
変換した入力値の重み付き和を計算します。
# 重みパラメータを初期化 w_mk = np.random.uniform(-10.0, 10.0, size=(M, K)) print(np.round(w_mk, 2)) # 重み付き和を計算 a_nk = np.dot(phi_x_nm, w_mk) print(np.round(a_nk[:5], 2))
[[-3.58 -1.01 -2.03]
[ 9.75 7.76 4.72]
[-8.36 8.14 -5.76]]
[[ 2.2 7.44 0.32]
[ 2.12 9.9 -0. ]
[ 1.48 8.86 -0.26]
[ 1.5 8.9 -0.25]
[ 1.13 10.52 -0.65]]
変換した入力値とロジスティック回帰のパラメータ$\mathbf{w} = (w_1, \cdots, w_K)$の内積を計算します。
この例では、重みパラメータw_mkの初期値を一様乱数で設定します。
重み付き和をソフトマックス関数で変換します。
# ソフトマックス関数による変換 y_nk = softmax(a_nk) print(np.round(y_nk[:5], 2))
[[0.01 0.99 0. ]
[0. 1. 0. ]
[0. 1. 0. ]
[0. 1. 0. ]
[0. 1. 0. ]]
始めに作成したsoftmax()で計算します。
出力$\mathbf{Y} = \{\mathbf{y}_1, \cdots, \mathbf{y}_N\}$の各項は、$0 < y_{n,k} < 1$、$\sum_{k=1}^K y_{n,k} = 1$の値をとり、対応する入力$\mathbf{x}_n$のクラスの確率の推定値を表します。
重みパラメータを更新します。
# 学習率を指定 eta = 0.01 # 変数を初期化 nabla_E_mk = np.zeros((M, K)) # クラスごとに更新 for k in range(K): # 勾配を計算 nabla_E_mk[:, k] = np.dot(phi_x_nm.T, y_nk[:, [k]] - t_nk[:, [k]]).flatten() # 最急降下法によりパラメータを更新 w_mk[:, k] -= eta * nabla_E_mk[:, k] print(np.round(w_mk, 2))
[[-2.59 -2.62 -1.41]
[10.6 6.29 5.33]
[-8. 7.39 -5.37]]
$\mathbf{w}_k$に関する誤差関数の勾配を次の式で計算します。
学習率$\eta$を指定して、次の式でパラメータ$\mathbf{w}$を更新します。
以上が1回の試行で行う処理(計算)です。
・全体のコード
最急降下法によりパラメータの最尤解を計算します。
多項式基底関数Phi_polynomial2d()・多項式基底関数Phi_gauss2d・シグモイド基底関数Phi_sigmoid2d()を関数phi()として複製して利用します。
# 試行回数を指定 max_iter = 10000 # 基底関数の数を指定 M = 3 # 基底関数を指定 #phi = Phi_polynomial2d #phi = Phi_gauss2d phi = Phi_sigmoid2d # 基底関数により入力値を変換 phi_x_nm = phi(x_nd, M) # 学習率を指定 eta = 0.01 # 重みパラメータを初期化 w_mk = np.random.uniform(-10.0, 10.0, size=(M, K)) # 変数を初期化 nabla_E_mk = np.zeros((M, K)) # 推移の記録用の配列を作成 trace_w_arr = np.zeros((max_iter, M, K)) # 重みパラメータ trace_E_list = np.zeros(max_iter) # 負の対数尤度 # ニ最急降下法による推定 for i in range(max_iter): # 重み付き和を計算 a_nk = np.dot(phi_x_nm, w_mk) # ソフトマックス関数による変換 y_nk = softmax(a_nk) # クラスごとに更新 for k in range(K): # 勾配を計算 nabla_E_mk[:, k] = np.dot(phi_x_nm.T, y_nk[:, [k]] - t_nk[:, [k]]).flatten() # 最急降下法によりパラメータを更新 w_mk[:, k] -= eta * nabla_E_mk[:, k] # 負の対数尤度関数を計算 y_nk = softmax(np.dot(phi_x_nm, w_mk)) E_val = -np.sum(t_nk * np.log(y_nk + 1e-7)) # 値を記録 trace_w_arr[i] = w_mk.copy() trace_E_list[i] = E_val # 途中経過を表示 #print(i + 1)
試行回数max_iterを指定して、重みパラメータw_mの値を繰り返し更新します。
また、推移の確認用にパラメータをtrace_w_arrに、負の対数尤度(交差エントロピー誤差)をtrace_E_listに格納していきます。
交差エントロピー誤差は、次の式で計算します。
計算に利用するy_nkは、更新したパラメータw_mkを使って再計算しています(y_nkを2回計算しており明らかに非効率ですが、推定の流れの分かりやすさを優先しました)。誤差は、学習の推移を確認するためのもので、推定には必要ありません。
・推定結果の可視化
推定結果と収束過程をグラフで確認します。
推定したパラメータを使って回帰曲面を計算します。
# K個の回帰曲面を計算
phi_x_valsm = phi(x_points, M, x_nd)
a_valsk = np.dot(phi_x_valsm, w_mk)
y_valsk = softmax(a_valsk)
$x_1$軸と$x_2$軸の値x_pointsの各要素に対して次の計算を行い、各点ごとにクラス1となる確率(z軸の値)を求めます。
ただし、phi_x_nmの全ての要素を一度に処理するために$\boldsymbol{\Phi} \mathbf{w}_{\mathrm{Logistic}}$で計算します。
決定境界を作図します。
# K個の色を指定 color_list = ['royalblue', 'orange', 'darkturquoise'] # K個の決定境界を作図 plt.figure(figsize=(12, 9)) plt.contour(X1, X2, model_density.reshape(x_dims)) # データ生成分布 for k in range(K): # クラスkの観測データを描画 k_idx = t_n == k plt.scatter(x=x_nd[k_idx, 0], y=x_nd[k_idx, 1], color=color_list[k], label='class ' + str(k+1)) # クラスkの決定境界を描画 plt.contour(X1, X2, y_valsk[:, k].reshape(x_dims), colors=['white', color_list[k], 'white'], levels=[0.0, 0.5, 1.0]) plt.xlabel('$x_1$') plt.ylabel('$x_2$') plt.suptitle('Logistic Regression', fontsize=20) plt.title('iter:' + str(max_iter) + ', N=' + str(N) + ', E(w)=' + str(np.round(E_val, 3)), loc='left') plt.colorbar(label='t') plt.legend() plt.grid() plt.show()
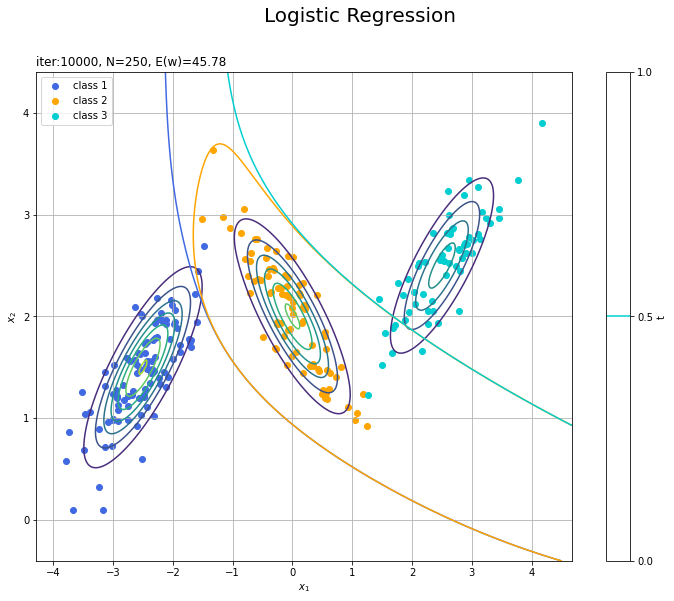
未知の入力$\mathbf{x}_{*}$に対して、$y_{*,k} > 0.5$であればクラス$k$($t_{*,k} = 1$)に分類します。そこで、クラスの境界線となる$y = 0.5$で決定境界(曲線)を引きます。
回帰曲面を作図します。
# クラス番号を指定 j = 0 # 回帰曲面を作図 fig = plt.figure(figsize=(12, 9)) ax = fig.add_subplot(projection='3d') # 3D用の設定 for k in range(K): k_idx = t_n == k if k == j: ax.scatter(xs=x_nd[k_idx, 0], ys=x_nd[k_idx, 1], zs=np.repeat(1.0, k_idx.sum()), c=color_list[k], label='class ' + str(k+1)) # クラスjの観測データ ax.scatter(xs=x_nd[k_idx, 0], ys=x_nd[k_idx, 1], zs=np.repeat(0.0, k_idx.sum()), facecolor='none', edgecolors=color_list[k], linestyles='--') # クラスjの観測データ:(底面) else: ax.scatter(xs=x_nd[k_idx, 0], ys=x_nd[k_idx, 1], zs=np.repeat(0.0, k_idx.sum()), c=color_list[k], label='class ' + str(k+1)) # クラスj以外の観測データ ax.plot_surface(X1, X2, y_valsk[:, j].reshape(x_dims), cmap='jet', alpha=0.5) # クラスjの回帰曲面 cntr = plt.contour(X1, X2, y_valsk[:, j].reshape(x_dims), colors=['white', color_list[j], 'white'], levels=[0.0, 0.5, 1.0], offset=0.5) # クラスjの決定境界 plt.contour(X1, X2, y_valsk[:, j].reshape(x_dims), colors=color_list[j], alpha=0.5, linestyles='--', levels=[0.5], offset=0.0) # クラスjの決定境界:(底面) plt.contour(X1, X2, model_density.reshape(x_dims), alpha=0.5, linestyles=':', offset=0.0) # データ生成分布:(底面) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.set_zlabel('$w_j^T \phi_j(x)$') fig.suptitle('Logistic Regression', fontsize=20) plt.title('$iter:' + str(max_iter) + ', N=' + str(N) + ', E(w)=' + str(np.round(E_val, 1)) + ', w_j=(' + ', '.join([str(w) for w in np.round(w_mk[: , j], 1)]) + ')$', loc='left') fig.colorbar(cntr, shrink=0.5, aspect=10, label='t') ax.legend() #ax.view_init(elev=90, azim=270) # 表示アングル plt.show()
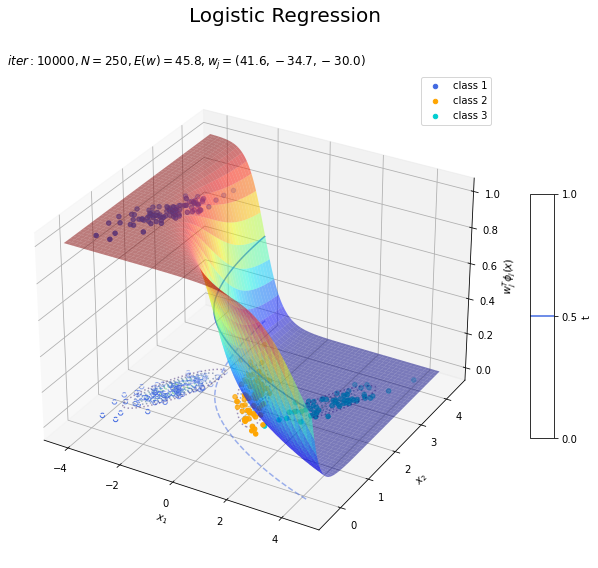
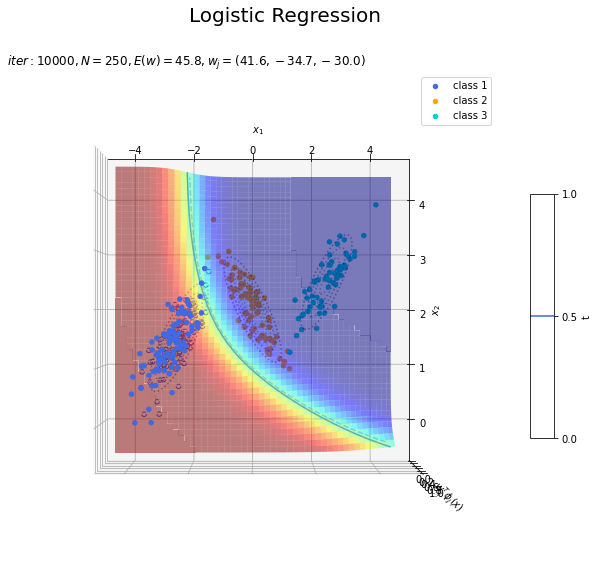
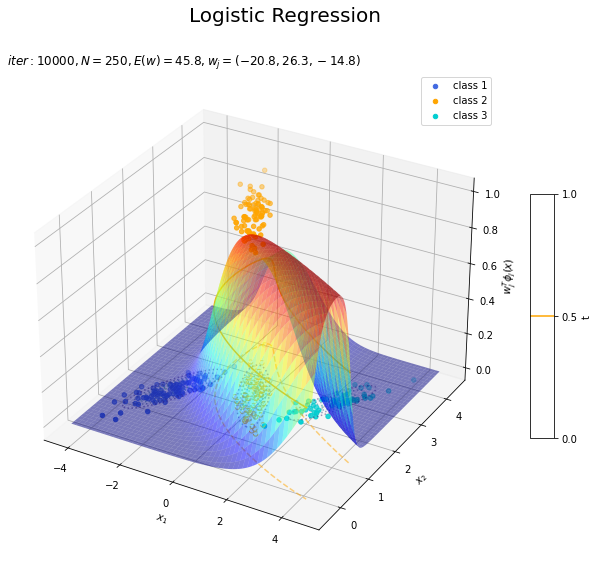
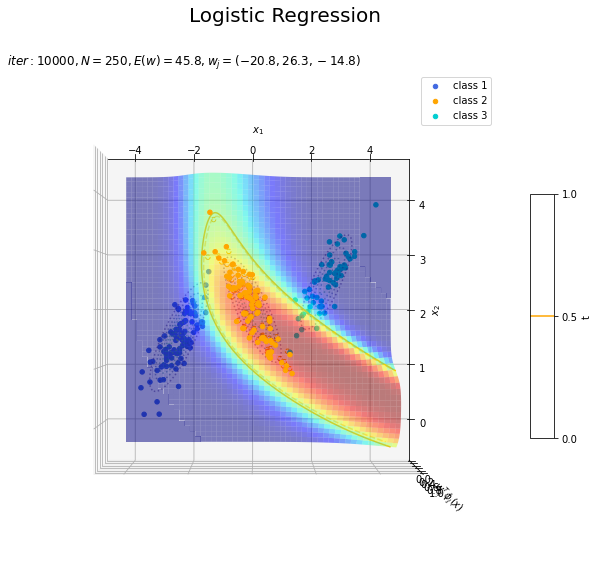
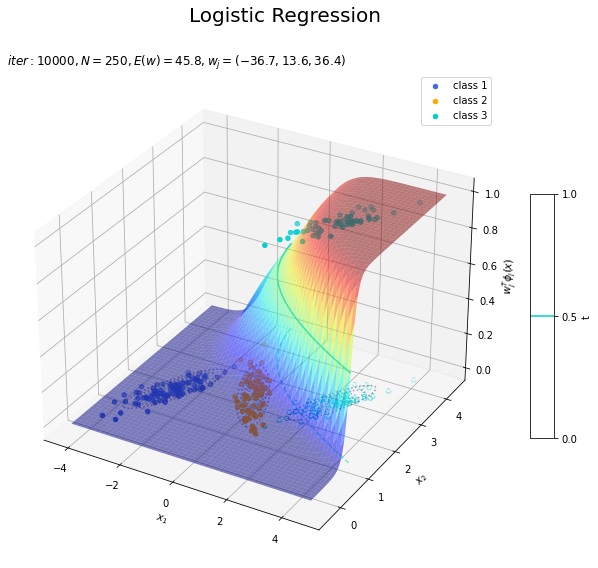
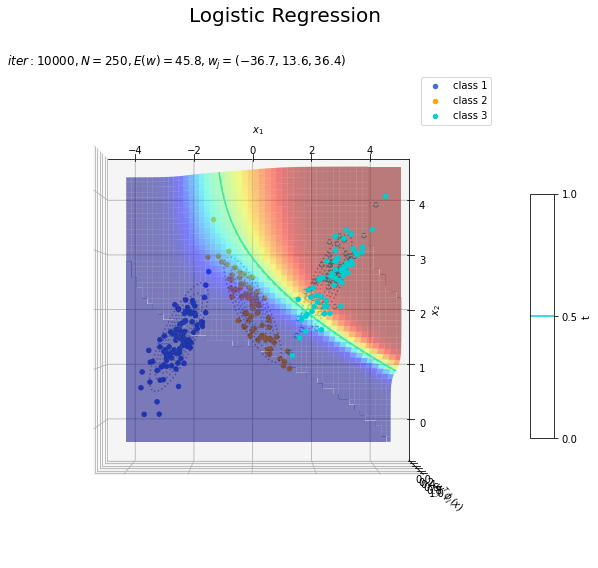
真上から見た図が、決定境界の図に対応します。
収束する過程をアニメーションで確認します。
決定境界の推移を作図します。
・作図コード(クリックで展開)
# フレーム数を指定 frame_num = 100 # 1フレーム当たりの試行回数を計算 iter_per_frame = max_iter // frame_num # 図を初期化 fig = plt.figure(figsize=(12, 9)) fig.suptitle('Logistic Regression', fontsize=20) # 作図処理を関数として定義 def update(i): # 前フレームのグラフを初期化 plt.cla() # i回目のパラメータを取得 w_mk = trace_w_arr[i*iter_per_frame] E_val = trace_E_list[i*iter_per_frame] # K個の回帰曲面を計算 a_valsk = np.dot(phi_x_valsm, w_mk) y_valsk = softmax(a_valsk) # 決定境界を作図 plt.contour(X1, X2, model_density.reshape(x_dims)) # データ生成分布 for k in range(K): # クラスkの観測データを描画 k_idx = t_n == k plt.scatter(x=x_nd[k_idx, 0], y=x_nd[k_idx, 1], color=color_list[k], label='class ' + str(k + 1)) # クラスkの決定境界を描画 plt.contour(X1, X2, y_valsk[:, k].reshape(x_dims), colors=['white', color_list[k], 'white'], levels=[0.0, 0.5, 1.0]) plt.xlabel('$x_1$') plt.ylabel('$x_2$') plt.title('iter:' + str(i*iter_per_frame+1) + ', E(w)=' + str(np.round(E_val, 3)), loc='left') plt.legend() plt.grid() # gif画像を作成 anime_logistic = FuncAnimation(fig, update, frames=frame_num, interval=100) # gif画像を保存 anime_logistic.save('ch4_3_4_LogisticRegression_cntr.gif')
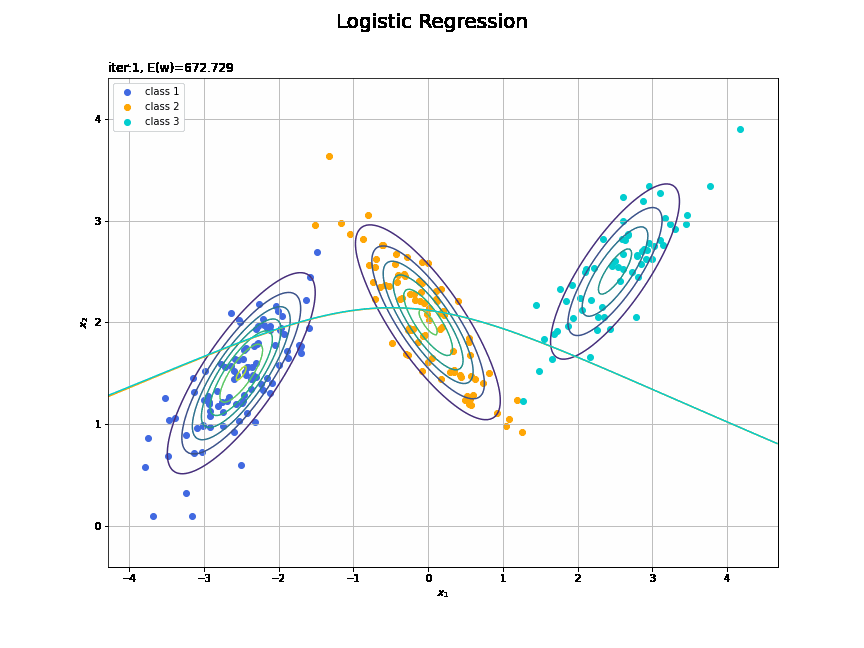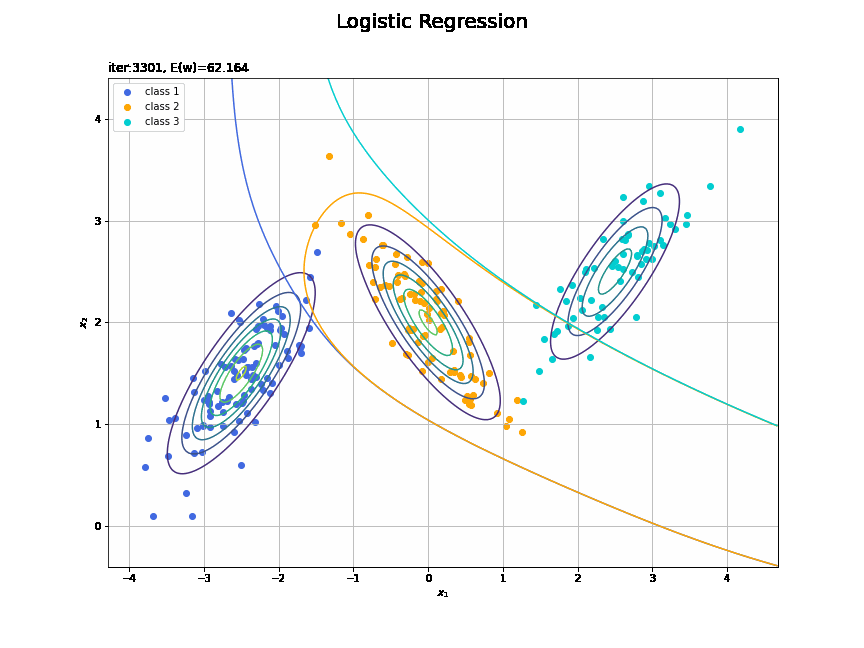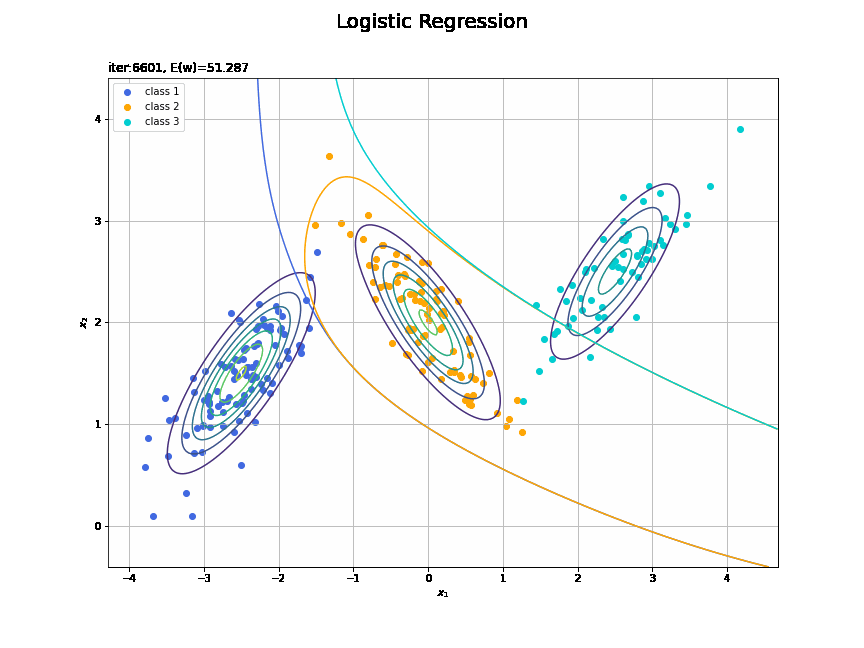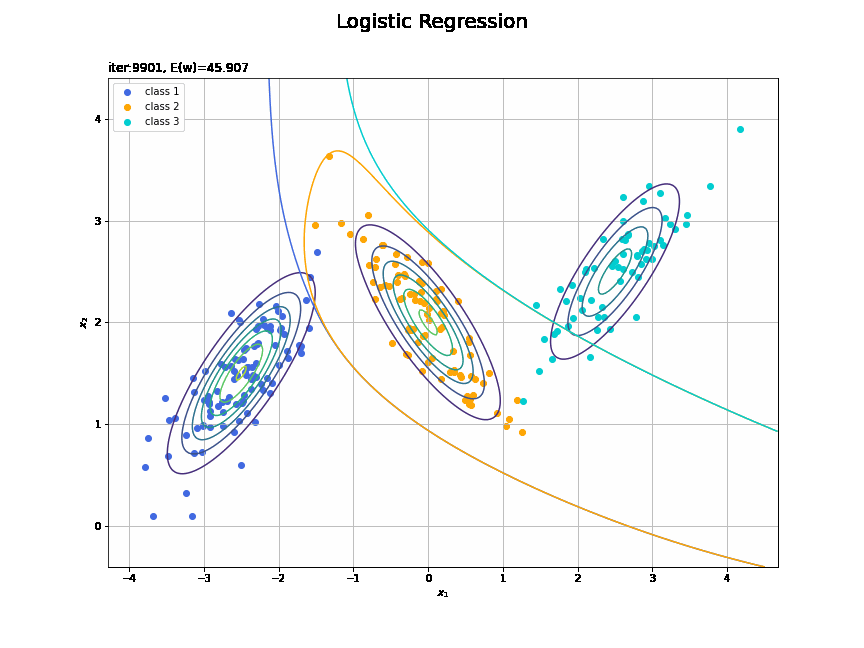
回帰曲面の推移を作図します。
・作図コード(クリックで展開)
# フレーム数を指定 frame_num = 100 # 1フレーム当たりの試行回数を計算 iter_per_frame = max_iter // frame_num # クラス番号を指定 j = 0 # 図を初期化 fig = plt.figure(figsize=(9, 8)) ax = fig.add_subplot(projection='3d') # 3D用の設定 fig.suptitle('Logistic Regression', fontsize=20) # 作図処理を関数として定義 def update(i): # 前フレームのグラフを初期化 plt.cla() # i回目のパラメータを取得 w_mk = trace_w_arr[i*iter_per_frame] E_val = trace_E_list[i*iter_per_frame] # K個の回帰曲面を計算 a_valsk = np.dot(phi_x_valsm, w_mk) y_valsk = softmax(a_valsk) # 回帰曲面を作図 for k in range(K): k_idx = t_n == k if k == j: ax.scatter(xs=x_nd[k_idx, 0], ys=x_nd[k_idx, 1], zs=np.repeat(1.0, k_idx.sum()), c=color_list[k], label='class ' + str(k+1)) # クラスjの観測データ ax.scatter(xs=x_nd[k_idx, 0], ys=x_nd[k_idx, 1], zs=np.repeat(0.0, k_idx.sum()), facecolor='none', edgecolors=color_list[k], linestyles='--') # クラスjの観測データ:(底面) else: ax.scatter(xs=x_nd[k_idx, 0], ys=x_nd[k_idx, 1], zs=np.repeat(0.0, k_idx.sum()), c=color_list[k], label='class ' + str(k+1)) # クラスj以外の観測データ ax.plot_surface(X1, X2, y_valsk[:, j].reshape(x_dims), cmap='jet', alpha=0.5) # クラスjの回帰曲面 plt.contour(X1, X2, y_valsk[:, j].reshape(x_dims), colors=['white', color_list[j], 'white'], levels=[0.0, 0.5, 1.0], offset=0.5) # クラスjの決定境界 plt.contour(X1, X2, y_valsk[:, j].reshape(x_dims), colors=color_list[j], alpha=0.5, linestyles='--', levels=[0.5], offset=0.0) # クラスjの決定境界:(底面) plt.contour(X1, X2, model_density.reshape(x_dims), alpha=0.5, linestyles=':', offset=0.0) # データ生成分布:(底面) ax.set_xlabel('$x_1$') ax.set_ylabel('$x_2$') ax.set_zlabel('$w_j^T \phi_j(x)$') ax.set_title('$iter:' + str(i*iter_per_frame + 1) + ', E(w)=' + str(np.round(E_val, 1)) + ', w_j=(' + ', '.join([str(w) for w in np.round(w_mk[: , j], 1)]) + ')$', loc='left') ax.legend() #ax.view_init(elev=0, azim=315) # 表示アングル:(横から) #ax.view_init(elev=90, azim=270) # 表示アングル:(上から) # gif画像を作成 anime_logistic = FuncAnimation(fig, update, frames=frame_num, interval=100) # gif画像を保存 anime_logistic.save('ch4_3_4_LogisticRegression2D_srfc_1.gif')
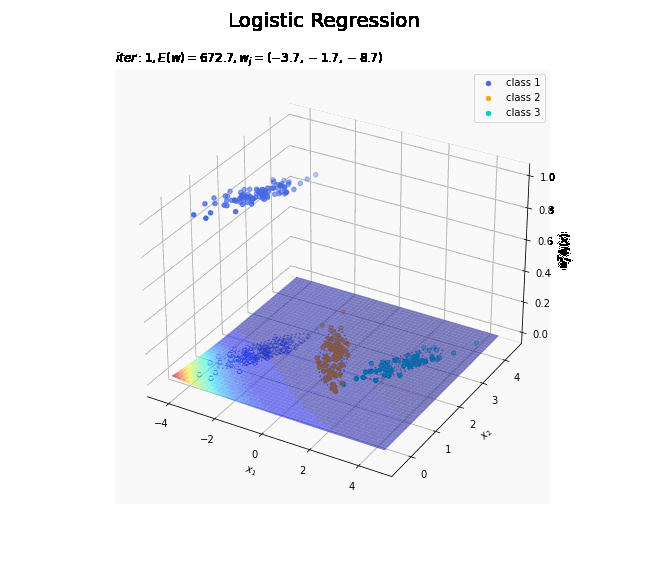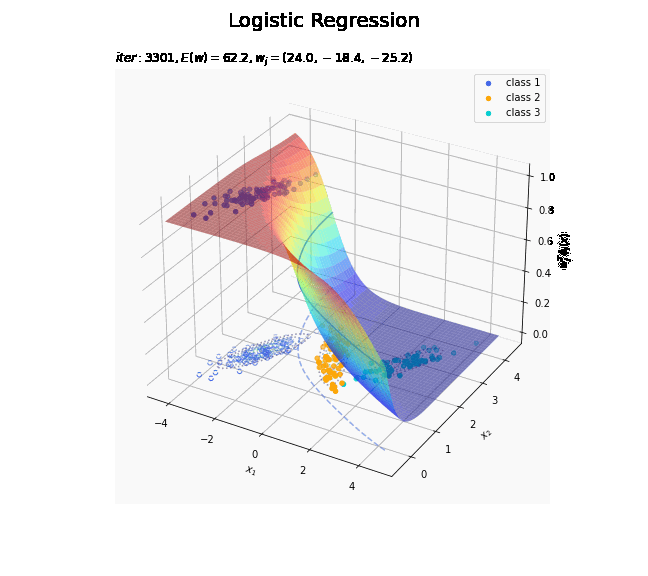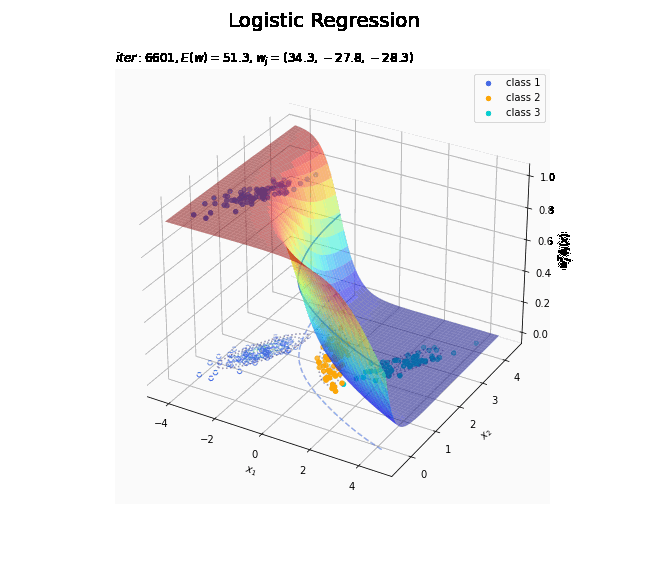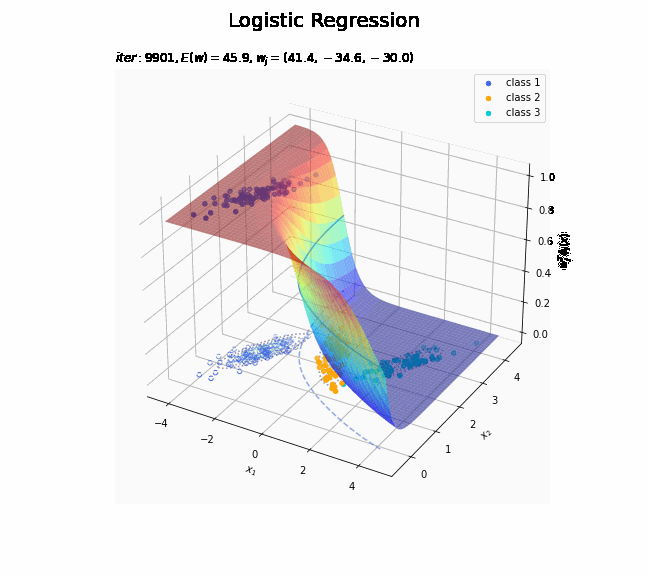
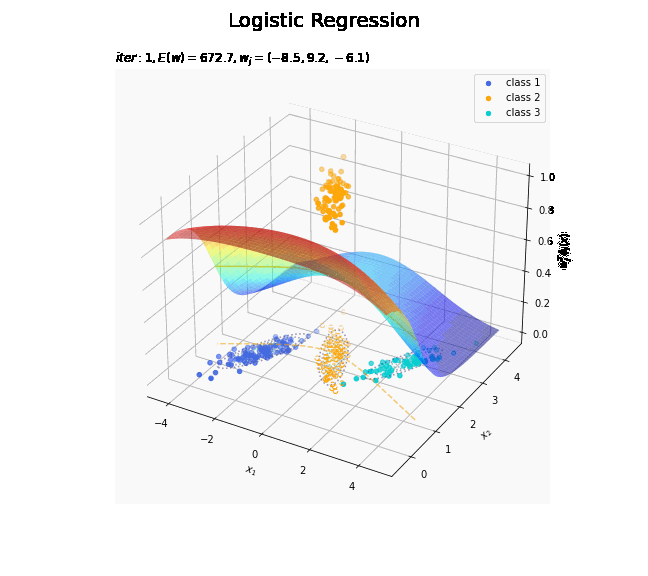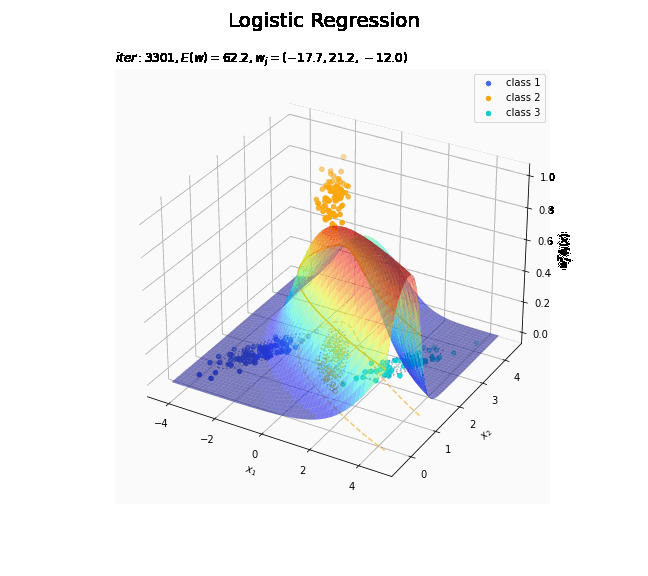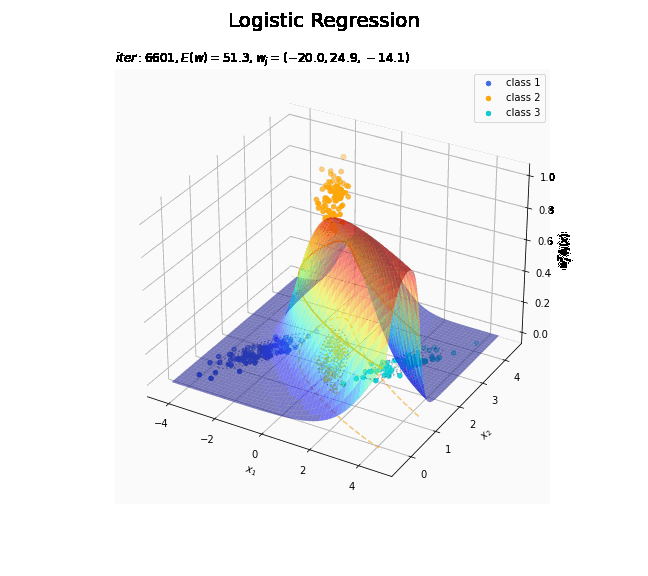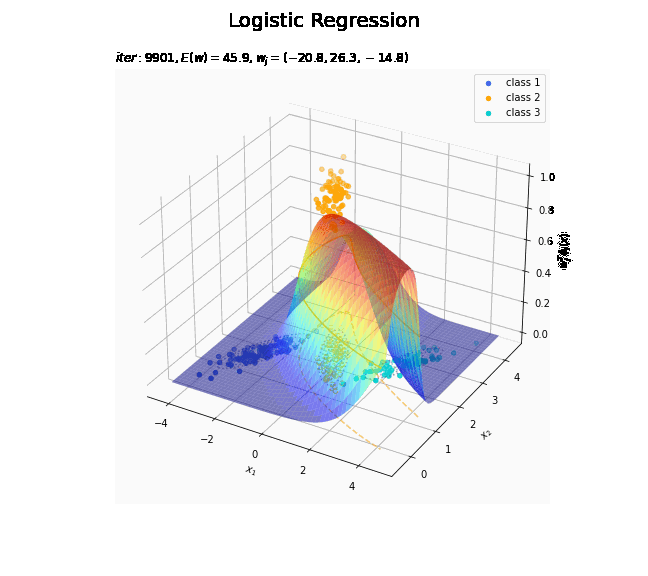
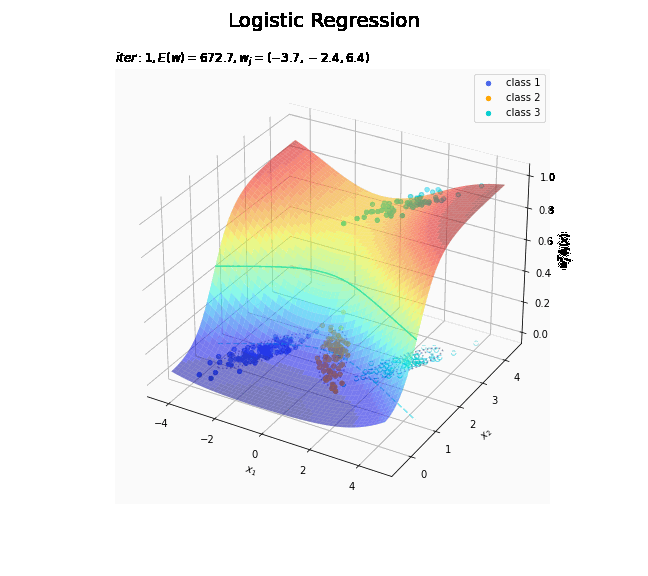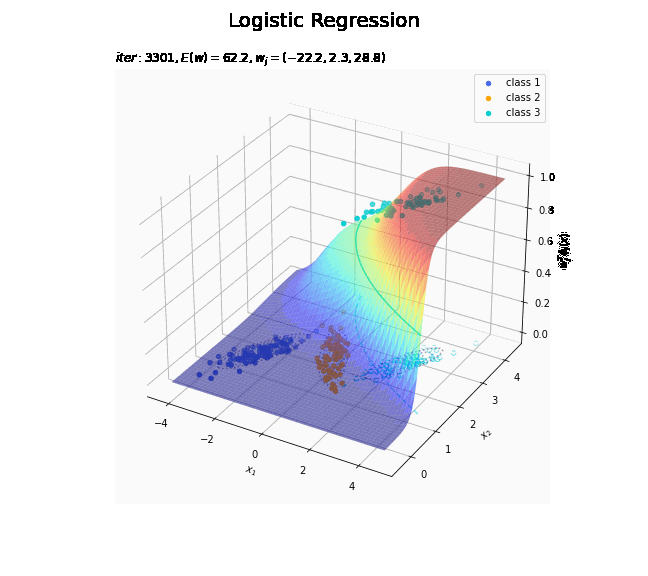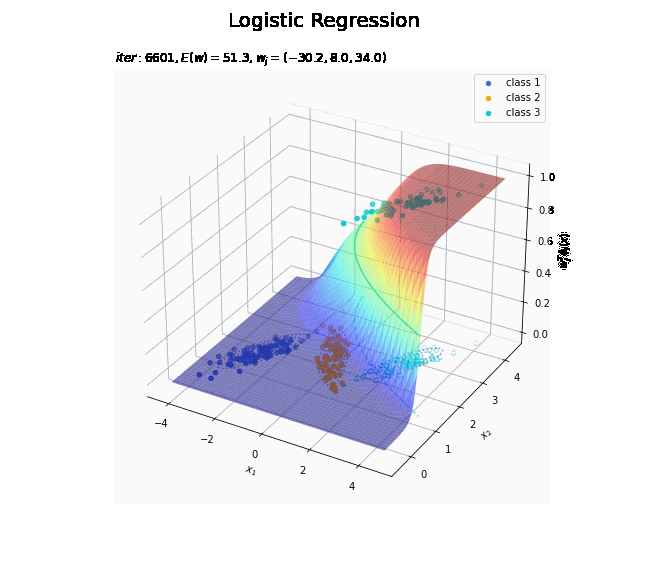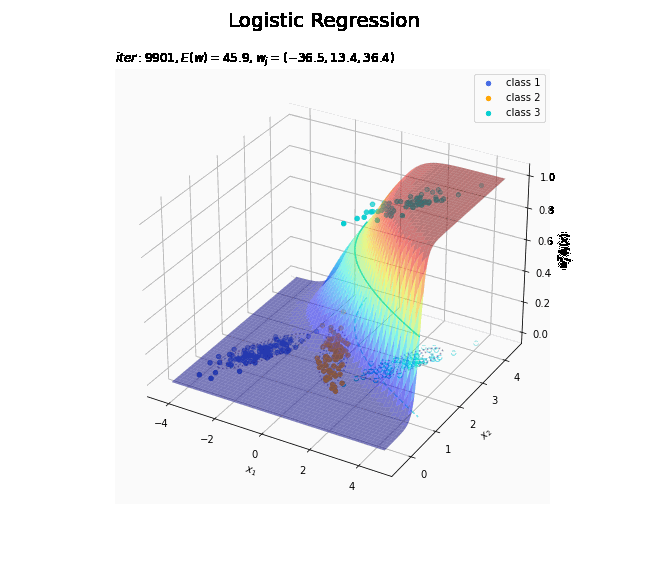
徐々にデータに適合していくのを確認できます。
誤差の推移のグラフを作成します。
# 誤差の推移を作図 plt.figure(figsize=(12, 9)) plt.plot(np.arange(1, max_iter+1), trace_E_list) plt.xlabel('iteration') plt.ylabel('E(w)') plt.suptitle('Cross-Entropy Error', fontsize=20) plt.title('$E(w^{(' + str(max_iter) + ')})=' + str(np.round(trace_E_list[max_iter-1], 3)) + '$', loc='left') plt.grid() plt.show()
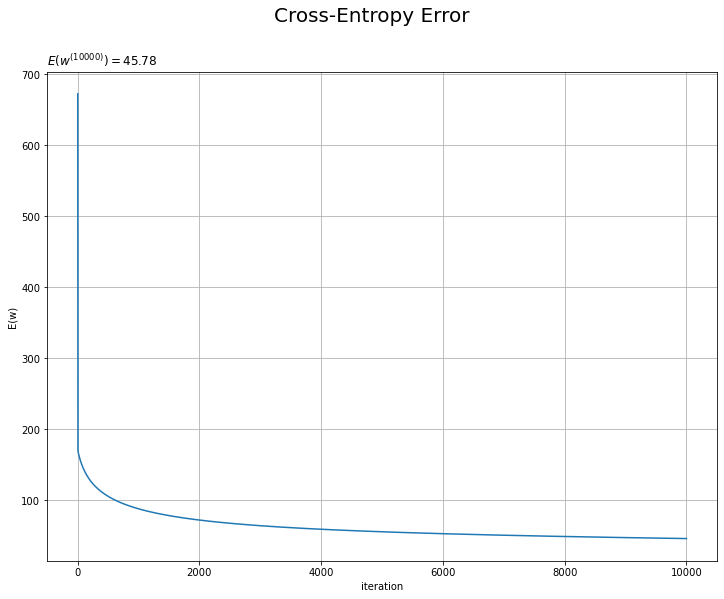
試行回数が増えるにしたがって誤差が小さくなっているのを確認できます。交差エントロピー誤差(負の対数尤度関数)の値が下がるのは、尤度関数の値が上がるのを意味します。
重みパラメータの推移のグラフを作成します。
# 重みの推移を作図 plt.figure(figsize=(12, 9)) for m in range(M): for k in range(K): plt.plot(np.arange(1, max_iter+1), trace_w_arr[:, m, k], label='$w_{' + str(m+1) + str(k+1) + '}$') plt.xlabel('iteration') plt.ylabel('$w_m$') plt.suptitle('Parameter', fontsize=20) plt.title('$w^{(' + str(max_iter) + ')}=(' + ', '.join([str(w) for w in np.round(trace_w_arr[max_iter-1], 2)]) + ')$', loc='left') plt.legend() plt.grid() plt.show()
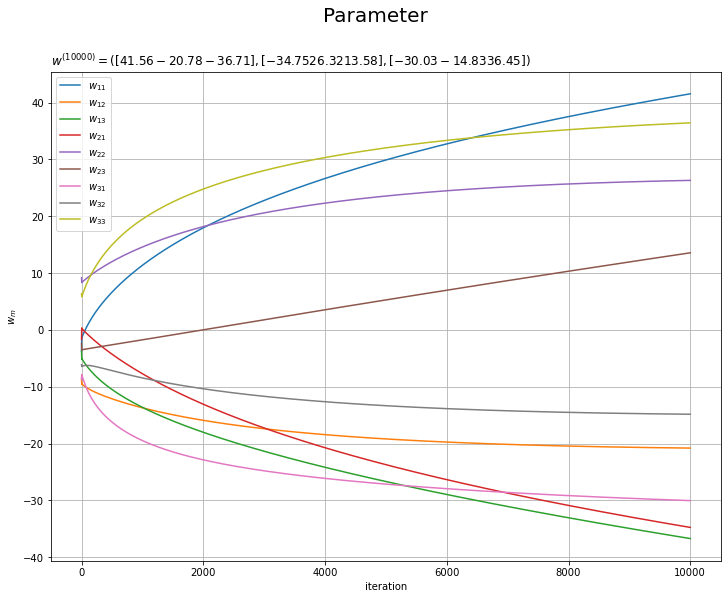
以上で、多クラスロジスティック回帰の最尤推定を実装と結果の可視化ができました。
参考文献
- C.M.ビショップ著,元田 浩・他訳『パターン認識と機械学習 上下』,丸善出版,2012年.
おわりに
他の節と同じくニュートン法でやりたかったのですが、組めませんでした。多クラスの場合のヘッセ行列の計算と値の持ち方が謎です。
2022年3月6日は、Berryz工房・カントリーガールズ・Buono!の元メンバー嗣永桃子さんのお誕生日です♪
引退後にハマったので生でパフォーマンスを観たことが無いのですが、ライブを観てみたかった。
【次節の内容】
つづく